Chapitre 7 Galerie de graphiques avec ggplot
Cette section contient des exemples de visualisations obtenues avec la librairie ggplot2.
Elle reprend des exemple de cette page https://r-statistics.co/Top50-Ggplot2-Visualizations-MasterList-R-Code.html adaptés en français et augmentés d’explications
Les exemples seront réalisés avec le jeu de données des manchots de la librairie palmerpenguins, dont vous pouvez avoir un aperçu avec la fonction summary().
library(palmerpenguins) # charche le jeu de données 'penguins'
data("penguins")
summary(penguins) # résumé des variables du dataframe## species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g sex year
## Adelie :152 Biscoe :168 Min. :32.10 Min. :13.10 Min. :172.0 Min. :2700 female:165 Min. :2007
## Chinstrap: 68 Dream :124 1st Qu.:39.23 1st Qu.:15.60 1st Qu.:190.0 1st Qu.:3550 male :168 1st Qu.:2007
## Gentoo :124 Torgersen: 52 Median :44.45 Median :17.30 Median :197.0 Median :4050 NA's : 11 Median :2008
## Mean :43.92 Mean :17.15 Mean :200.9 Mean :4202 Mean :2008
## 3rd Qu.:48.50 3rd Qu.:18.70 3rd Qu.:213.0 3rd Qu.:4750 3rd Qu.:2009
## Max. :59.60 Max. :21.50 Max. :231.0 Max. :6300 Max. :2009
## NA's :2 NA's :2 NA's :2 NA's :27.1 Lien entre deux variables quantitatives
Pour examiner le lien qu’il peut exister entre deux variables (quantitatives) d’une population , le plus courant est de réaliser un nuage de points (scatterplot in english). Sur la base de la forme de ce nuage, on pourra décider de calculer la corrélation, faire une régression etc.
7.1.1 Nuage de points simple
library(palmerpenguins) # charche le jeu de données 'penguins'
data("penguins")
simpleplot <- ggplot(data = penguins, aes(x=body_mass_g, y=flipper_length_mm))+
geom_point()
simpleplot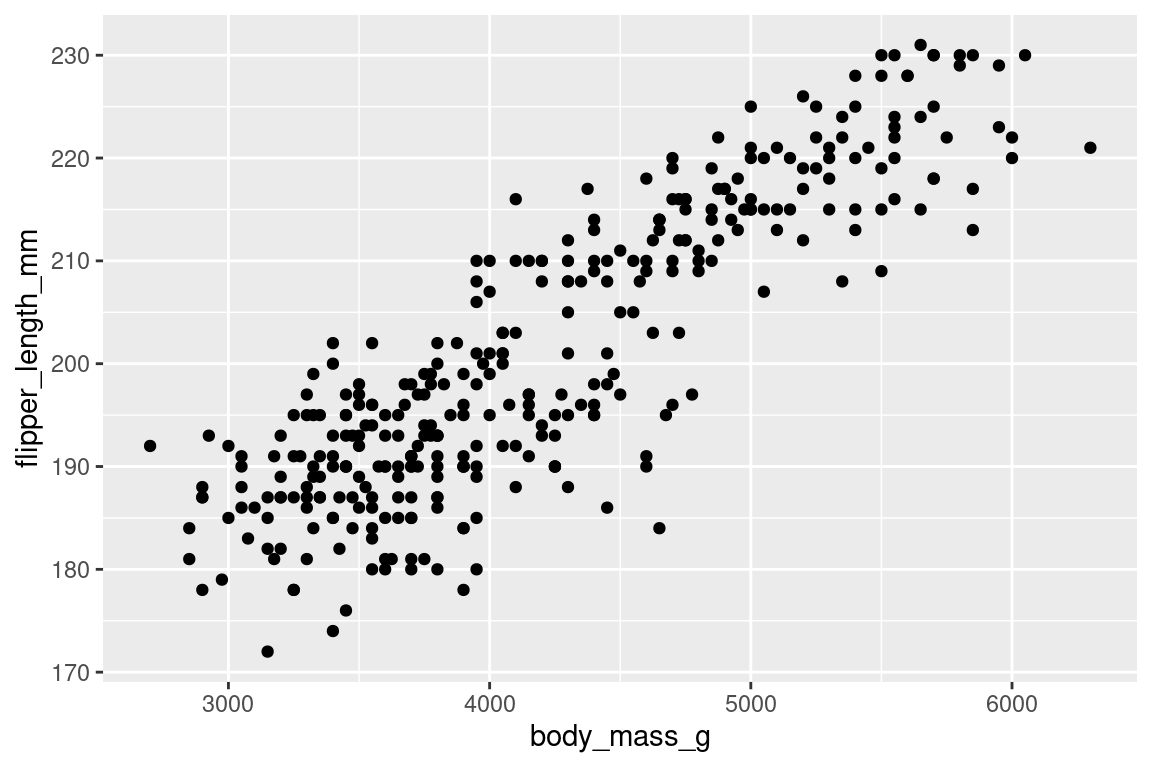
Beaucoup de choses sont faites par défaut avec ce code minimal :
- figuré des points (cercles pleins )
- figurés sont de couleurs noire
- thème graphique par défaut : grille blanche sur fond gris clair
- axes étiquetés avec le nom brut des variables (axe X et Y)
- graduations des axes
- pas de titre
Je préfère utiliser un thème moins marqué, theme_light, qu’on peut ajouter à l’objet simpleplot, pour changer de thème.
simpleplot_light <- ggplot(data = penguins, aes(x=body_mass_g, y=flipper_length_mm))+
geom_point()+
theme_light()
simpleplot_light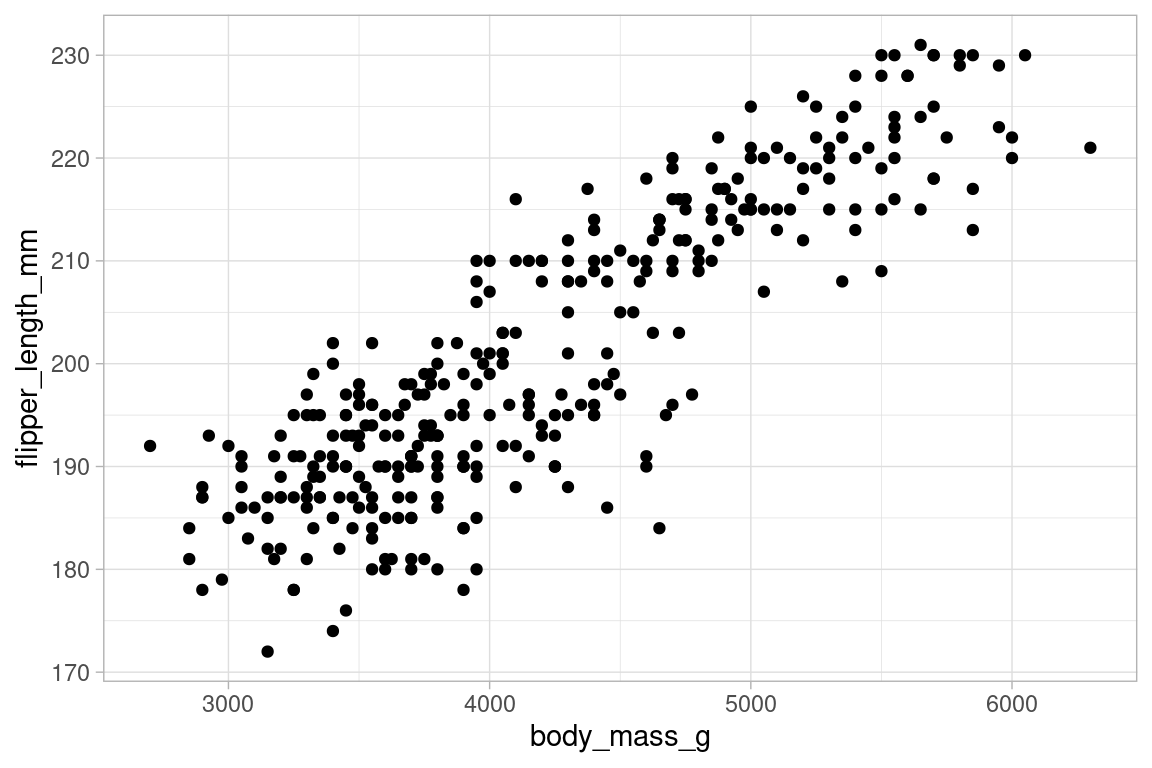
Pour modifier la couleur , on utilise les arguments de la fonction geom_point.
On utilise un nom de la liste des couleurs prédéfinies de R, un entier entre 0 et 25 pour la forme des figurés:
simpleplot_light <- ggplot(data = penguins, aes(x=body_mass_g, y=flipper_length_mm))+
geom_point(color="darkcyan", shape=18)+
theme_light()
simpleplot_light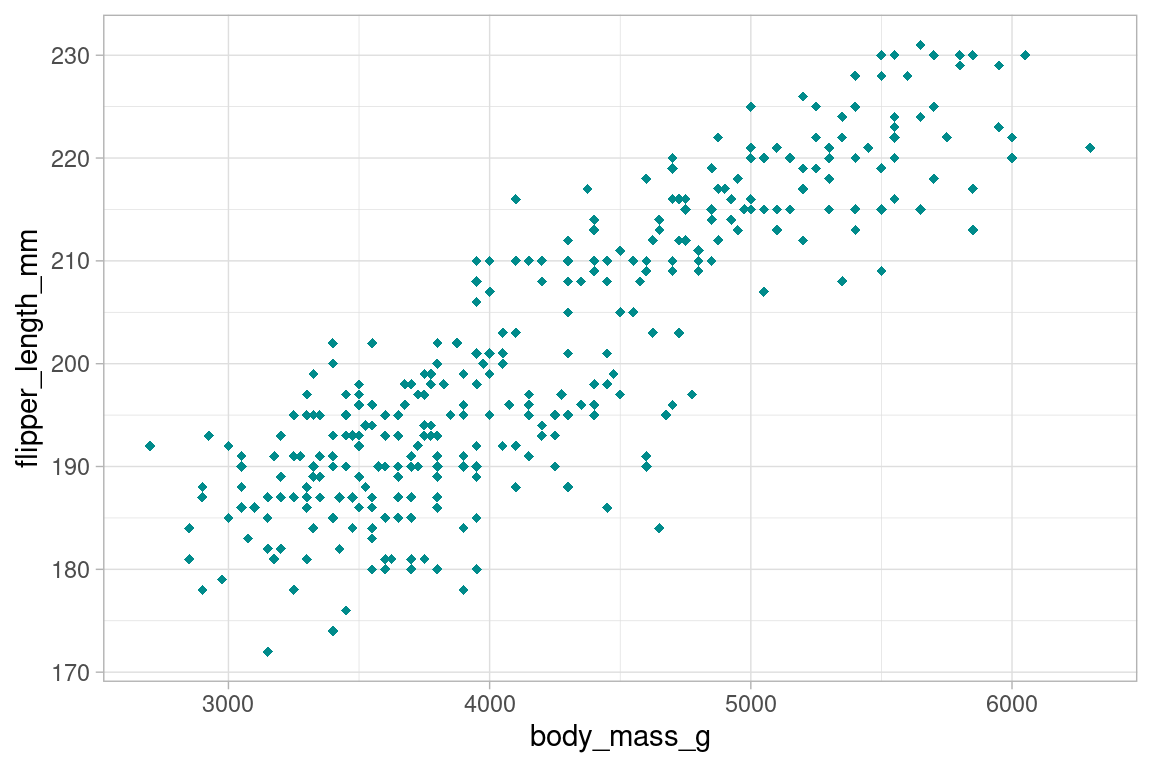
Beaucoup d’autres options sont modifiables, pour en avoir un aperçu, tapez la commandes vignette("ggplot2-specs") dans la console R.
7.1.2 Ajouter un titre , étiqueter les axes
On continue de “décorer” l’objet ggplot avec des fonctions spécifiques :
labs()pour le titre, le sous-titre -xlabetylabpour les étiquettes des axes X et Y- évenutellement,
caption, un argument de la fonctionlabs()pour une sorte de cartouche, idéale pour citer les sources.
plot_complet <- ggplot(data = penguins, aes(x=body_mass_g, y=flipper_length_mm))+
geom_point(color="darkcyan", shape=18)+
theme_light()+
labs(title = "titre principal du graphique",subtitle = "un sous-titre ", caption = "Source: dataset palmerpenguins ")+
xlab("étiquette des abscisses")+
ylab("étiquette des ordonnées ")
plot_complet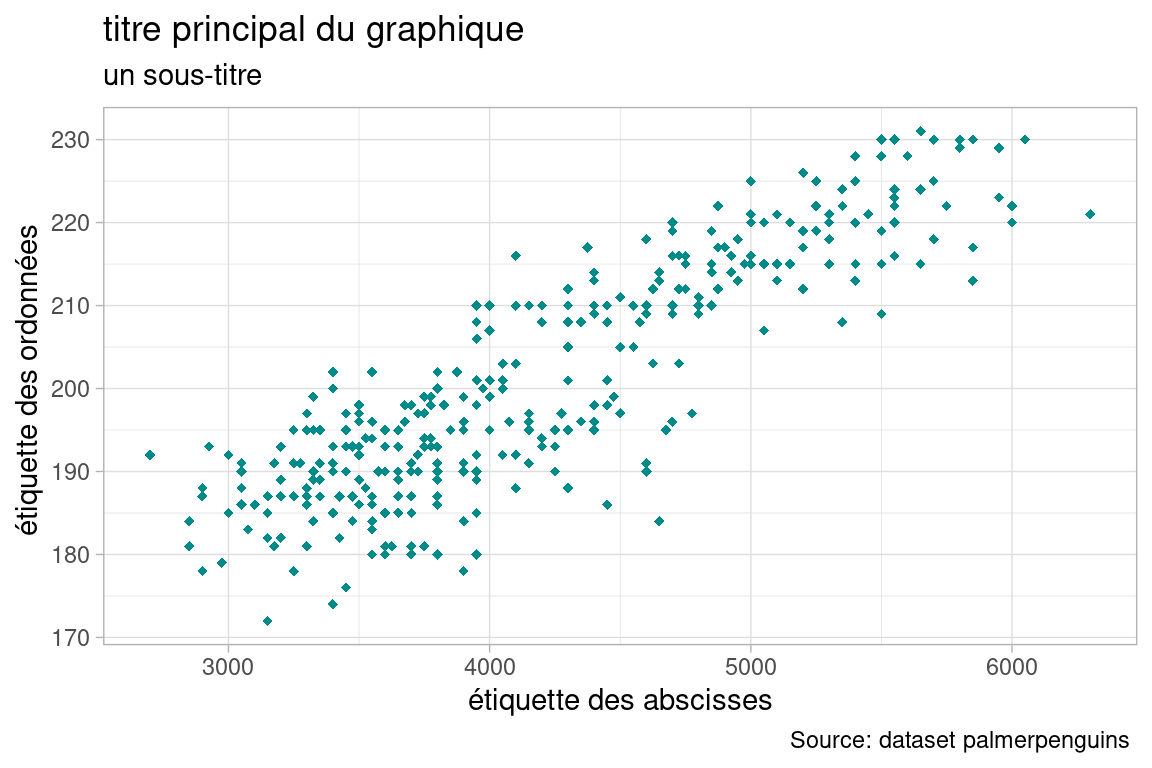
7.1.3 Nuage de points colorés par variable continue (quantitative)
Pour colorer les figurés selon une troisième variable, on demande à ggplot de réaliser un mapping (i.e. on “câble” deux variables ensemble) entre une variable du dataframe , et une variable visuelle.
Cela se fait à l’intérieur de la fonction geom_point(), avec l’expression aes(color=bill_length_mm).
Cette expression réalise la liaison entre la valeur de la variable bill_length_mm et la couleur du figuré (argument color). L’échelle de couleur par défaut est un gradient de nuances de bleu.
Ici nous allons lier la variable continue bill_length_mm avec la couleur des figurés.
plot_color_by_var <- ggplot(data = penguins, aes(x=body_mass_g, y=flipper_length_mm))+
geom_point(aes(color=bill_length_mm))+
theme_light()+
labs(title = "Masse des manchots en fonction de la longueur des nageoires",subtitle = "coloré par la longueur du bec (mm)", caption = "Source: dataset palmerpenguins ")+
xlab("masse du corps (g)")+
ylab("longueur des nageoires (mm) ")
plot_color_by_var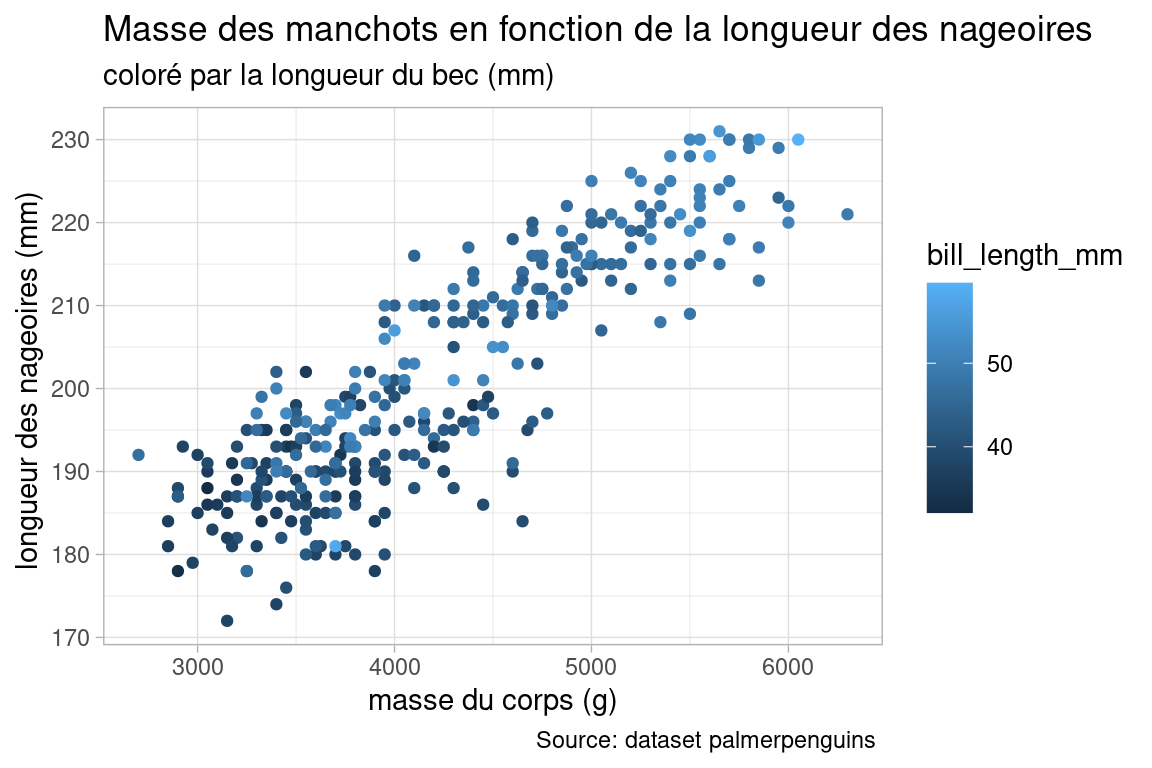
7.1.4 Nuage de points colorés par variable discrète (qualitative)
Le principe est similaire au précédent, seule la variable affectée à la couleur va changer, on utilise cette fois la variable qualitative species. GGplot2 se charge du reste et affecte une couleur à chaque espèce dans le nuage de points.
plot_color_by_var <- ggplot(data = penguins, aes(x=body_mass_g, y=flipper_length_mm))+
geom_point(aes(color=species))+
theme_light()+
labs(title = "Masse des manchots en fonction de la longueur des nageoires",subtitle = "coloré par espèce", caption = "Source: dataset palmerpenguins ")+
xlab("masse du corps (g)")+
ylab("longueur des nageoires (mm) ")
plot_color_by_var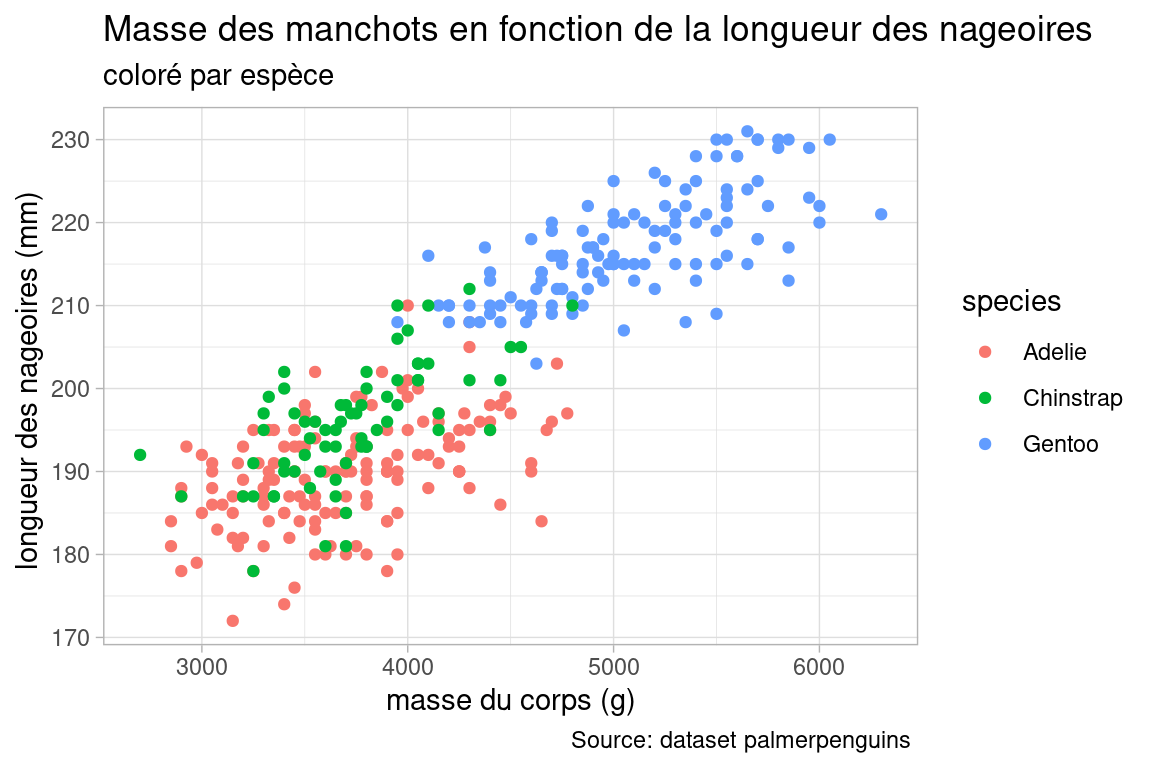
7.1.5 Nuage de points et droite de regression / de lissage
plot_color_by_var <- ggplot(data = penguins, aes(x=body_mass_g, y=flipper_length_mm))+
geom_point()+
geom_smooth(method="lm", se=F)+
theme_light()+
labs(title = "Masse des manchots en fonction de la longueur des nageoires",subtitle = "avec la droite de regression", caption = "Source: dataset palmerpenguins ")+
xlab("masse du corps (g)")+
ylab("longueur des nageoires (mm) ")
plot_color_by_var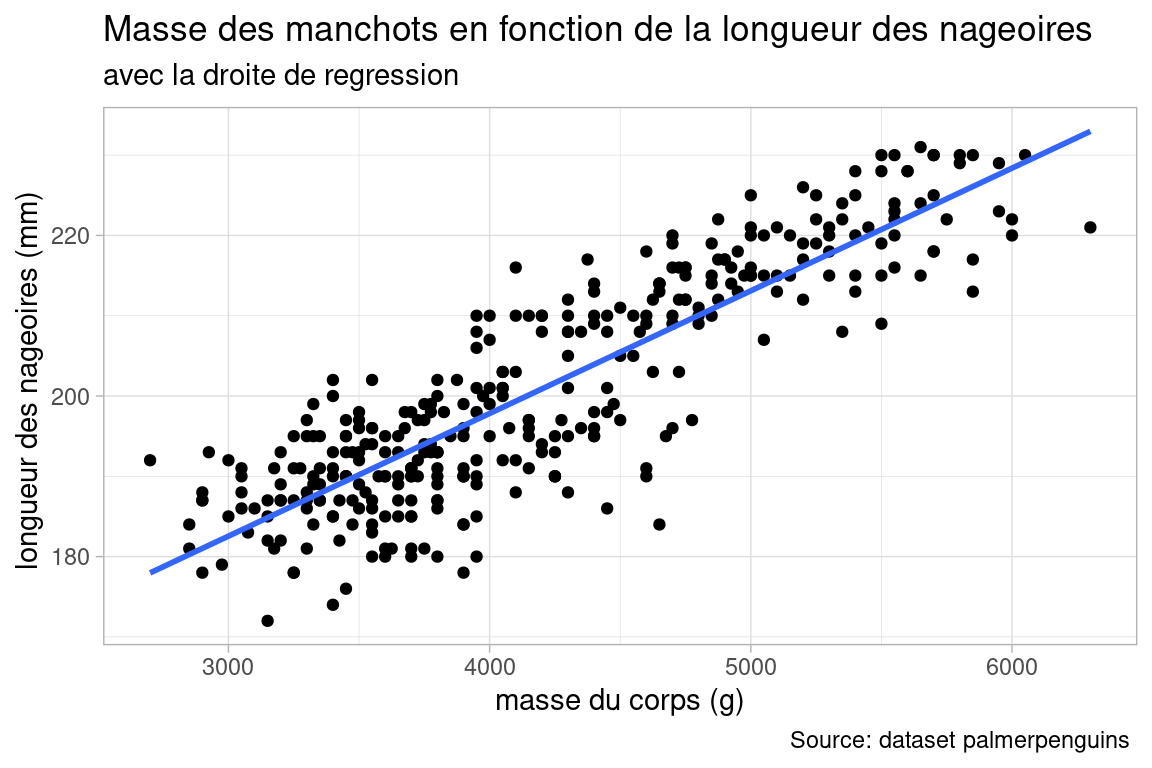
GGplot propose une fonction de lissage, geom_smooth, qui propose des courbes qui généralisent le nuage de points, par différentes méthodes. La plus simple est la droite de régression linéaire, mais il est également possible d’utiliser une LOESS ou une régression linéaire multiple.
On peut également afficher “l’intervalle de confiance” de la courbe, avec l’argument se de la fonction geom_smooth() mis à TRUE. C’est l’intervalle autour de la courbe dans lequel se trouveraient les autres droites de régression si on venait à répéter la régression sur un sous-échantillon un grand nombre de fois. Quand cet intervalle est étroit, on peut l’interpréter comme le signe d’une regression robuste , car 95% (la valeur par défaut, que l’on peut forcer avec l’argument level ) des droites se retrouveraient dans cette enveloppe, si on répétait l’opération sur d’autres échantillons de la même population.
7.1.6 À ne pas faire
Il est tentant d’utiliser toutes les variables visuelles et ajouts possibles, en un seul graphique : - couleur des figurés - forme des figurés - taille des figurés - transparence - étiquettes de certains points - droites/courbes de prédictions
le résultat final perd en lisibilité et le message est complexe : il faut alors tenir compte de plusieurs variables à la fois pour interpréter le graphique. Voici un exemple
library(viridis)
overcomplicated_plot <- ggplot(data = penguins %>% na.omit(), aes(x=body_mass_g, y=flipper_length_mm, group=species))+
geom_point(aes(color=species, shape=sex, size=bill_length_mm))+
geom_smooth(method="lm", se=T, aes(linetype=species), color="blue")+
theme_light()+
labs(title = "Graphique montrant trop de variables",subtitle = "Ne faites pas ça ", caption = "Source: dataset palmerpenguins ")+
xlab("masse du corps (g)")+
ylab("longueur des nageoires (mm) ")+
scale_colour_viridis_d()
overcomplicated_plot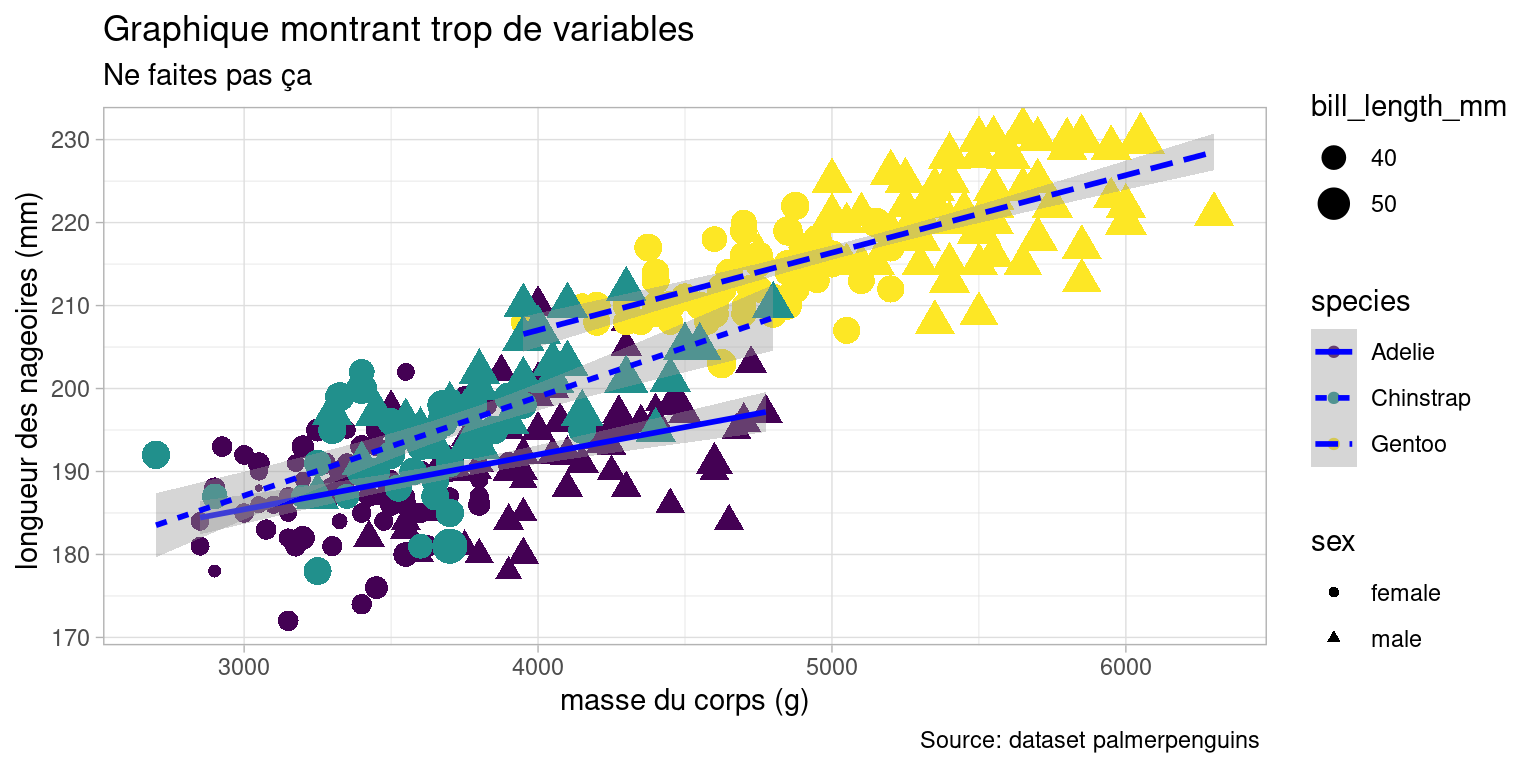
7.1.7 Ajouter les histogrammes marginaux
Parfois à cause de points superposés par exemple, ou masqués par la taille d’autres points voisins, il est difficile d’estimer visuellement la densité d’individus dans les zones de valeurs des deux variables X et Y. Il est pertinent d’ajouter , sur les côté du graphe, l’histogramme des variables choisies en X et Y.
On utilise pour ça le package ggExtra. Une fois le graphe ggplot initial constitué (ici margin_hist_plot), il est passé en argument à la fonction ggmarginal qui va le décorer avec les histogrammes des variables qui ont été affectées aux variables visuelles x et y de la fonction aes().
library(ggExtra)
margin_hist_plot <- ggplot(data = penguins %>% na.omit(), aes(x=body_mass_g, y=flipper_length_mm, group=species))+
geom_point(aes(color=species))+
theme_light()+
labs(title = "Masse et longueur des nageoires",subtitle = "Avec histogrammes marginaux", caption = "Source: dataset palmerpenguins ")+
xlab("masse du corps (g)")+
ylab("longueur des nageoires (mm) ")
ggMarginal(margin_hist_plot, type = "histogram", fill="transparent" )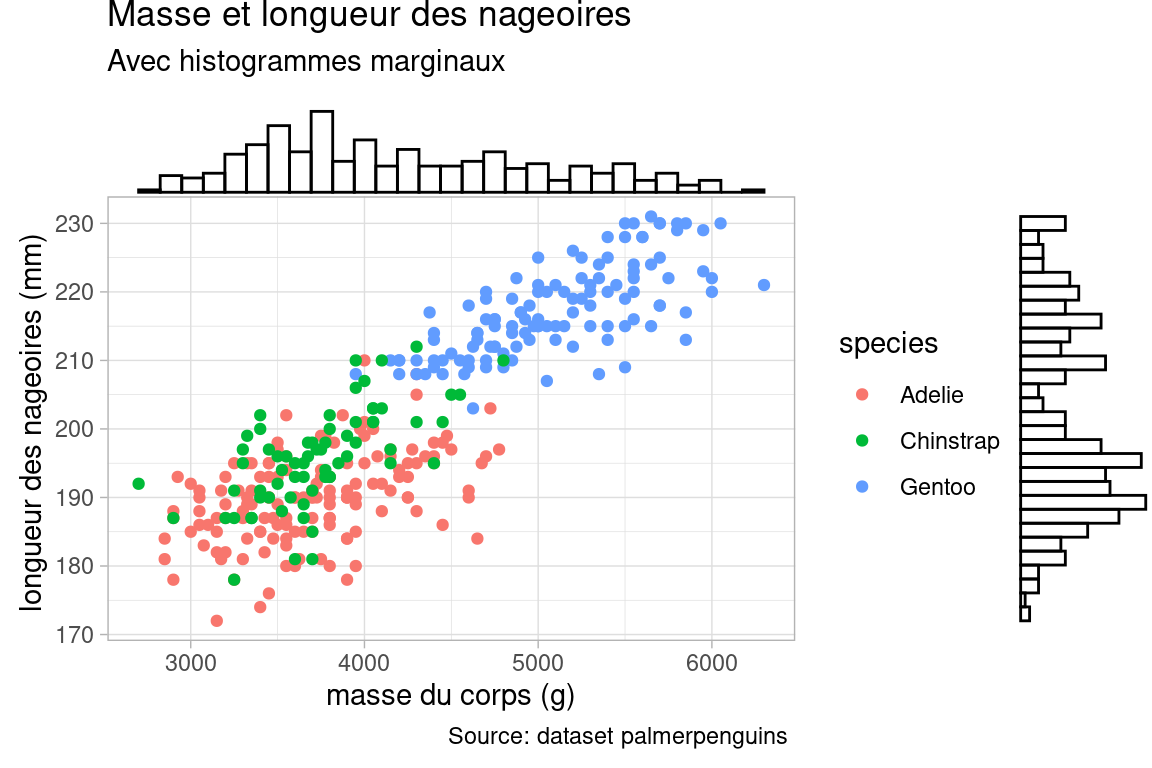
Attention cependant à ne pas surcharger le graphique inutilement, il faut que l’affichage des histogramme ait un intérêt (par exemple une bi-modalité) en lui même et qu’il puisse être mobilisé dans le discours/l’interprétation du graphique.
7.2 Lien entre plusieurs variables quantitatives
On s’intéresse maintenant à l’affichage simultané du lien qui peut exister entre plusieurs variables quantitatives. Souvent , ce lien est la corrélation, on appel donc ce genre de graphiques des correlogrammes
7.2.1 Correlogramme simple avec GGally
Le package GGally possède une fonction pour réaliser un corrélogramme, que l’on peut facilement compléter avec d’autres informatiosn utiles : distribution des variables individuellement, affichage du nuages de points etc…
on commence par filtrer les variables numériques du dataset des manchots à l’aide de la fonction select du package dplyr :
La fonction ggcorr réalise un corrélogramme simple, où la couleur représente la valeur du coefficient de corrélation sur une échelle de couleur bidirectionnelle.
library(GGally)
ggcorr(numeric_var_penguins,
name="corrélation de Pearson",
label=T,
layout.exp = 1.9)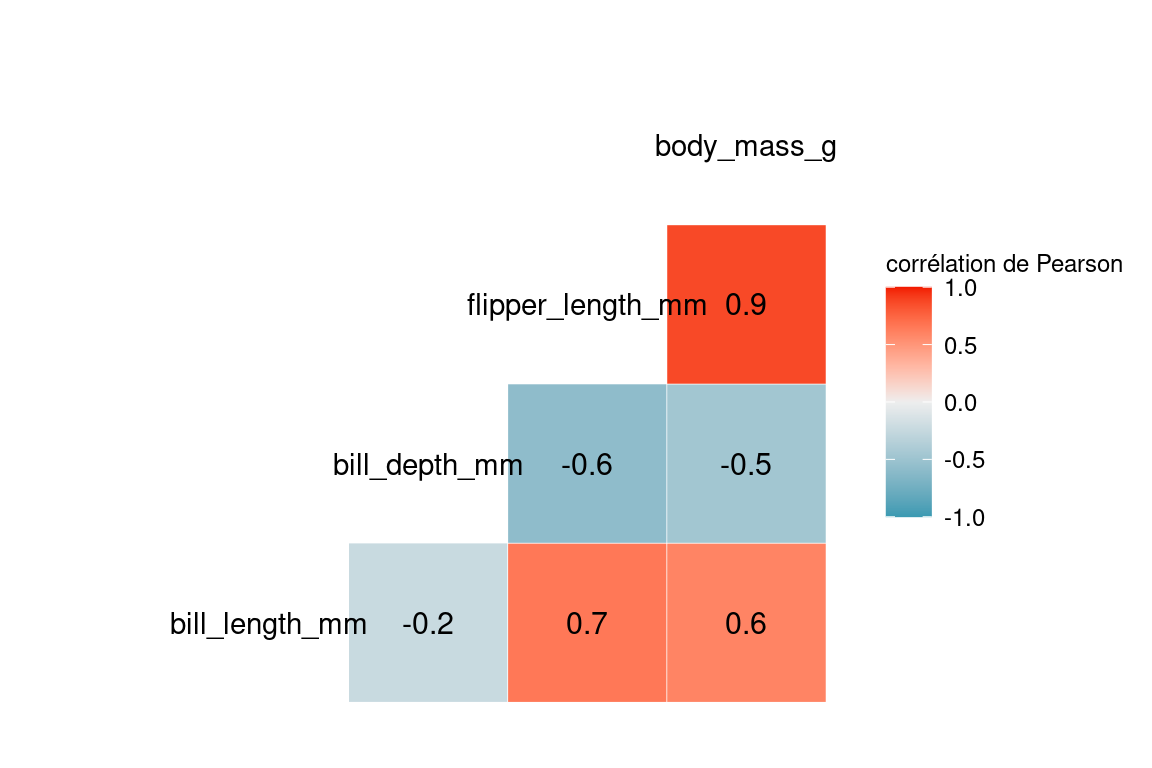
7.2.2 Correlogramme plus complexe avec GGally
On peut ajouter plusieurs autres informations utiles au corrélogramme à l’aide de la fonction ggpairs(). Voici un exemple avec notre jeu de données quantitatif sur les manchots :
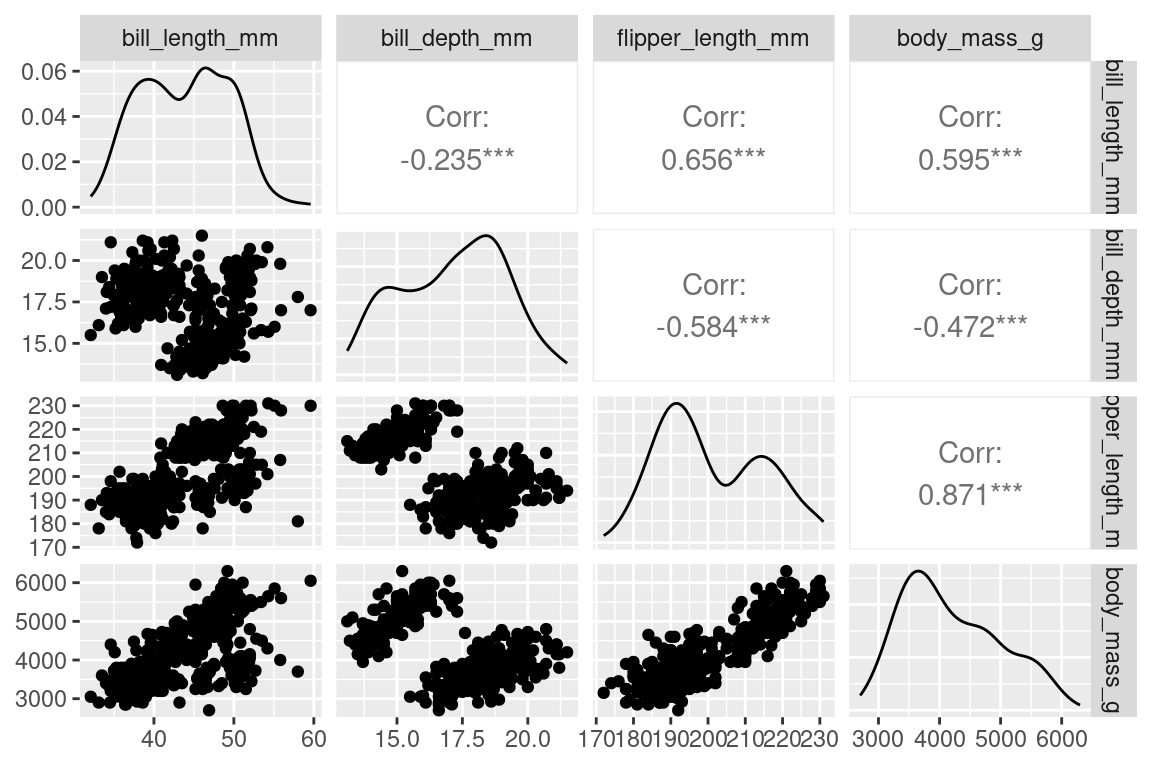
Plus généralement, la fonction ggcor() accepte des variables quantitatives et qualitatives. Dans le corrélogramme, à l’intersection de la colonne de la variable \(V1\) , et de la ligne de la variable \(V2\), et suivant leur type, on trouvera :
- Dans la matrice triangulaire inférieure :
- Un nuage de points , si \(V1\) et \(V2\) sont quantitatives
- des histogrammes par catégories si \(V1\) est qualitative et \(V2\) est quantitative
- Sur la diagonale :
- un histogramme si la variable est qualitative
- une densité si la variable est quantitative
- Dans la matrice triangulaire supérieure :
- le coefficient de correlation si \(V1\) et \(V2\) sont quantitatives avec leurs significativtés représentées par des asterisques (cf. la section 5.5 )
- des boxplots par catégorie si \(V1\) est qualitative et \(V2\) est quantitative
- une matrice de barplots, semblable à un mosaicplot, vu dans la section 5.7 dédiée au \(\chi^2\) ,quand \(V1\) et \(V2\) sont qualitatives
Les options de la fonction permettent de modifier l’affichage de différentes façons. Attention aux nombres de variables représentées, car on obtient vite des graphiques trop riches pour être lisibles, comme dans l’exemple ci-dessous
library(GGally)
ggpairs(penguins %>% select(-c(year)),
mapping = ggplot2::aes(color= species),
legend= 2,
upper = list(continuous = "cor", combo = "box_no_facet", discrete = "count", na =
"na"),
title ="Un corrélogramme trop riche, ne pas reproduire") +
theme_light()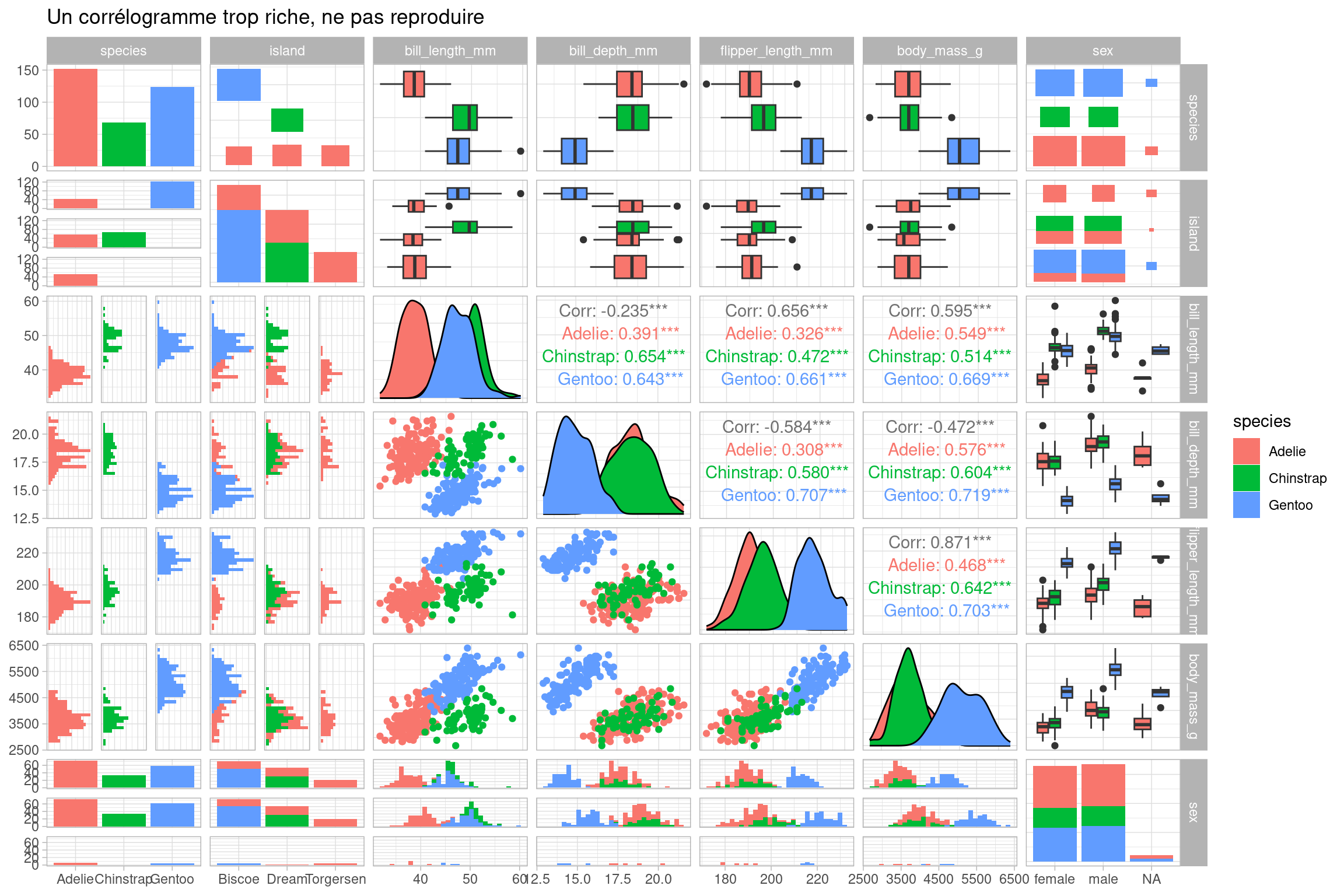
D’autres packages et exemples sont disponibles sur [https://r-graph-gallery.com/correlogram.html]
7.2.3 Parallel plot
Ce type de graphique est utilisé pour représenter les variables quantitatives comme des axes (souvent verticaux) parallèles, et les individus comme des lignes qui coupent ces axes au niveau des valeurs de leurs variables. On utilise la fonction ggparcoord du package GGally. Dans cet exemple, nous ne considérons que les variables quantitatives, avec l’argument columns.
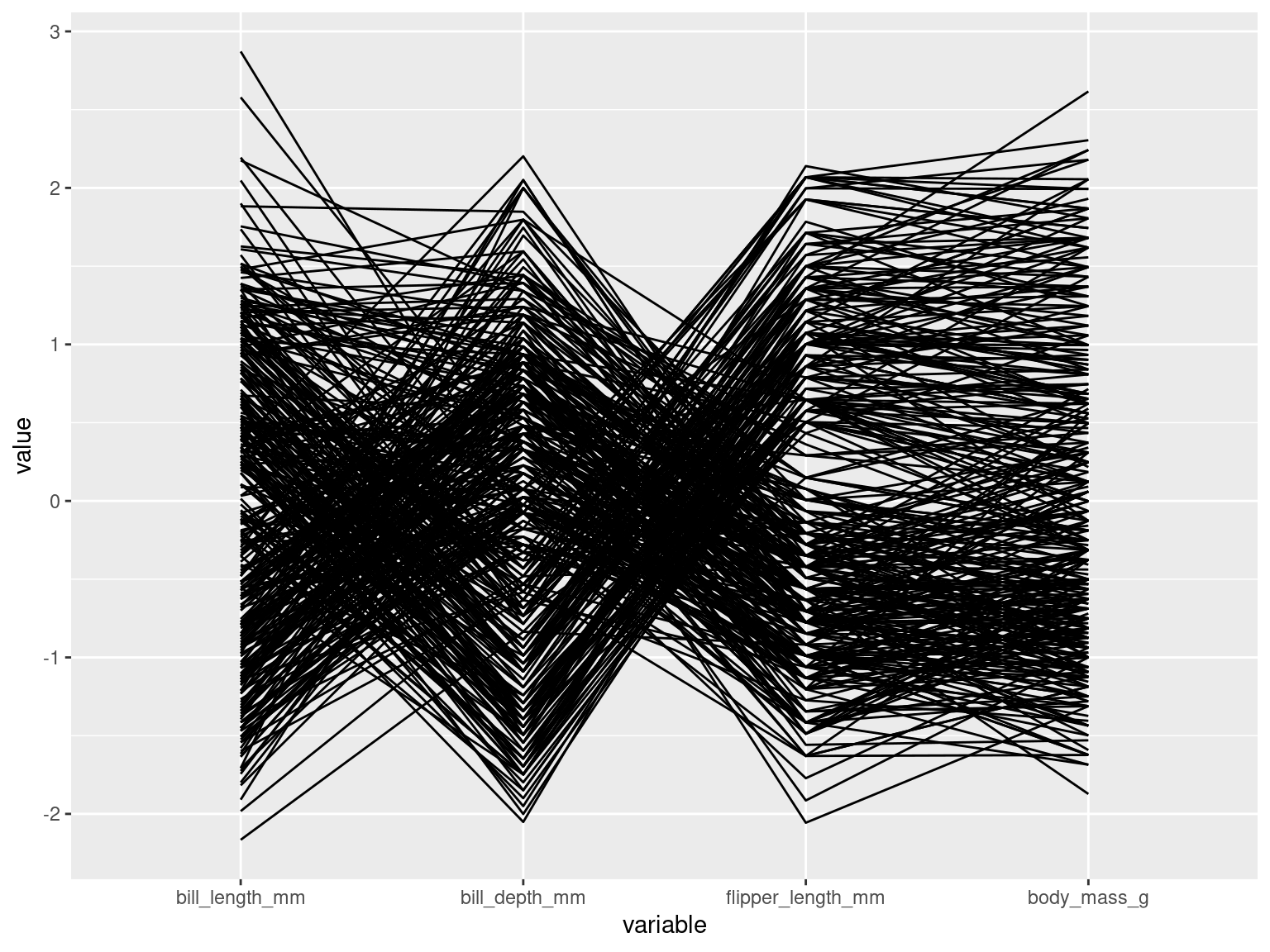
On peut constater que les variables ont été normalisées (on y a soustrait les moyennes et divisés par l’écart type) pour que les effets d’unité
Pour le moment ce graphique n’est pas satisfaisant , nous allons rendre plus transparentes les lignes des individus, et colorer par une variable qualitative, l’espèce.
ggparcoord(data=penguins,
columns = 3:6 ,# colonnes des variables quantitatives
alphaLines = 0.5, #transparence
groupColumn = "species",
) # couleur des groupes par espèce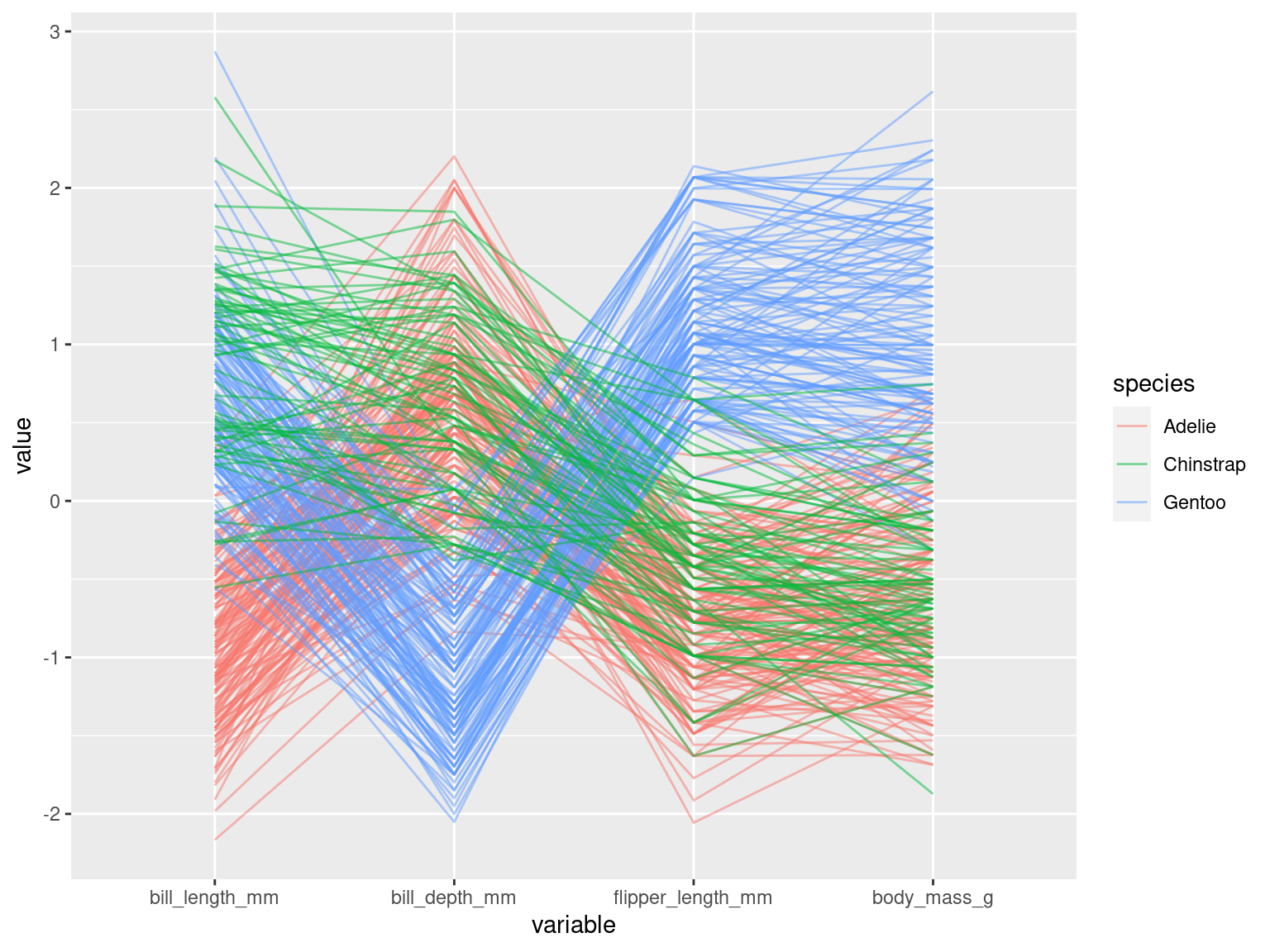
La couleur premet de voir que les valeurs qui décrivent la morphologie des manchots sont bien discriminées par leur espèce : les Gentoo sont de gros manchots à bec court.
Pour mettre en évidence un groupe particulier, on pourra par exemple modifier l’échelle de couleur, en mettant en gris le groupe qui ne nous intéresse pas, et en couleur le groupe qui nous intéresse :
ggparcoord(data=penguins,
columns = 3:6 ,# colonnes des variables quantitatives
alphaLines = 0.21, #transparence
groupColumn = "species",# couleur des groupes par espèce
)+
scale_color_manual(values=c( "purple2", "lightgray", "lightgray") ) # il y a trois espèces, on met en violet la première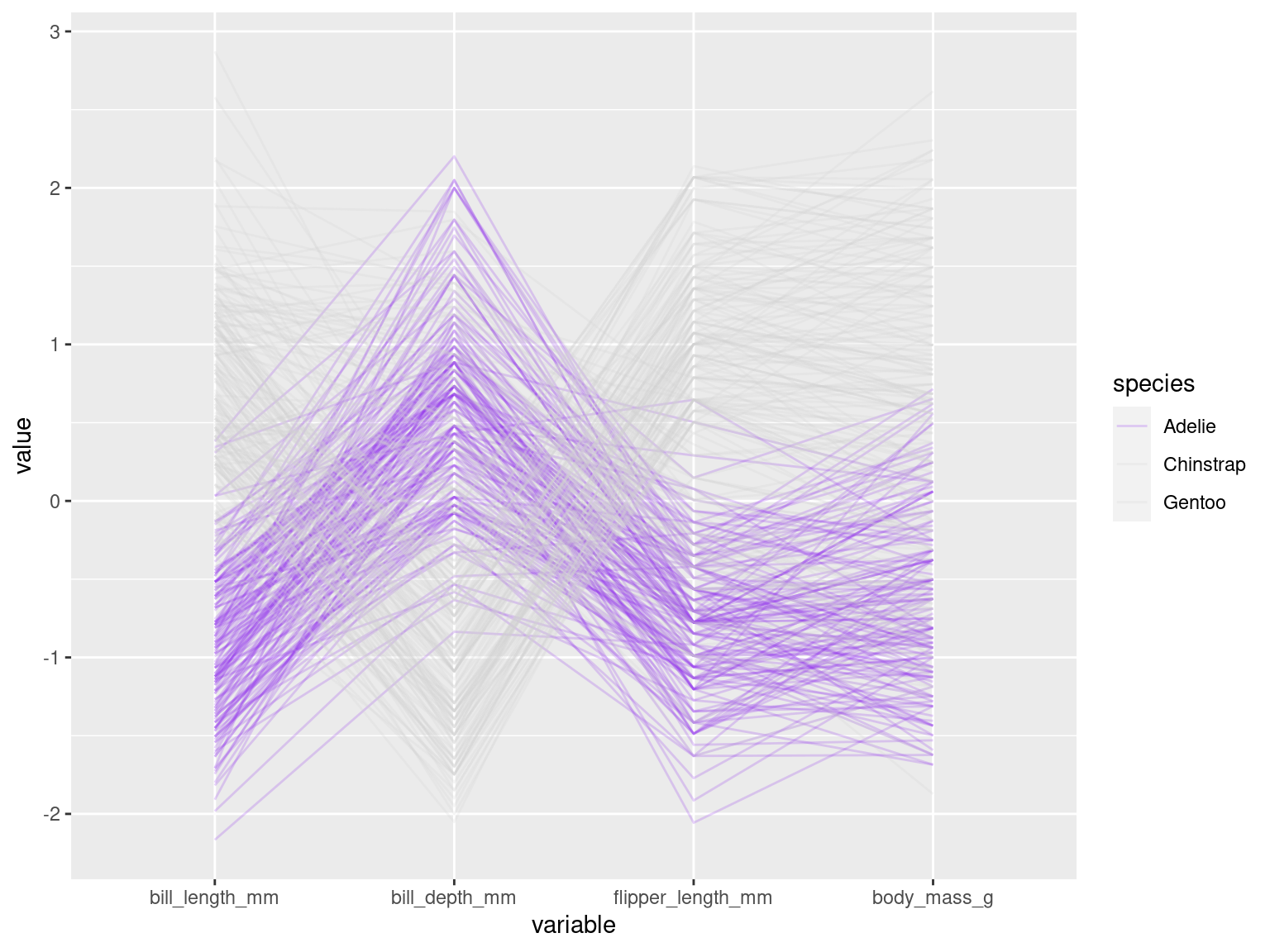
7.3 Lien entre deux variables qualitatives
Comme vu dans la section dédiée au test d’indépendance du \(\chi^2\), le lien entre deux variables qualitatives (ou modales ou factorielles) peut être visualisé au moyen de graphiques qui représentent tous plus ou moins la table de contingence des variables choisies
7.3.1 Mosaic plot simple
Un graphique “de base” peut être obtenu avec la fonction mosaicplot() qui prend comme argument une formule qui liste les varaibles qualitatives à croiser, sour la forme suivante : ~Variable1+Varibale2, comme dans l’exemple ci-dessous :
mosaicplot(~species+sex, data=penguins,main = "Moisaic plot simple", color=c("darkorange1", "royalblue1"))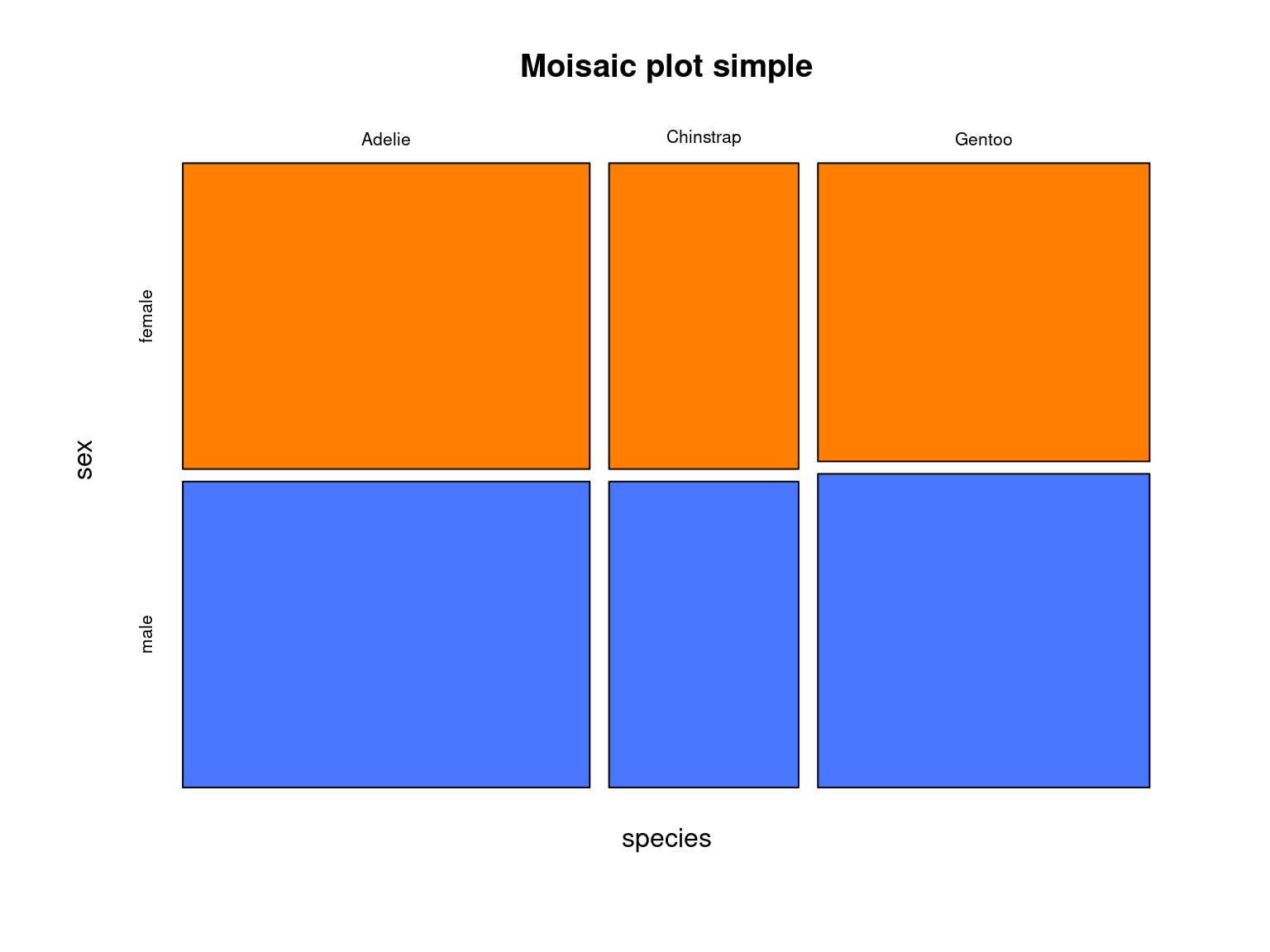
Les aires étant proportionnelles, cette visualisation met en évidence une proportion d’individus mâles (resp. femelles) invariante selon l’espèce.
7.3.2 Moisaic plot de ggplot2
A l’aide du package ggmosaic, on peut obtenir un graphique un peu plus atreyant, notamment en colorant facilement par une troisième variable, ce qui aura pour effet de diviser à nouveau les rectangles du mosaic plo, comme dans l’exemple ici, l’année.
library(ggmosaic)
library(viridis)
plot_mosaic <- ggplot(data=penguins %>% mutate(year=as.factor(year)) %>% na.omit) +
geom_mosaic(aes(x = product(species,sex), fill=year)) +
theme(panel.background = element_blank(),
panel.border = element_blank(),
panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
plot.background = element_blank(),
axis.ticks.y = element_blank(),
axis.ticks.x = element_blank())
plot_mosaic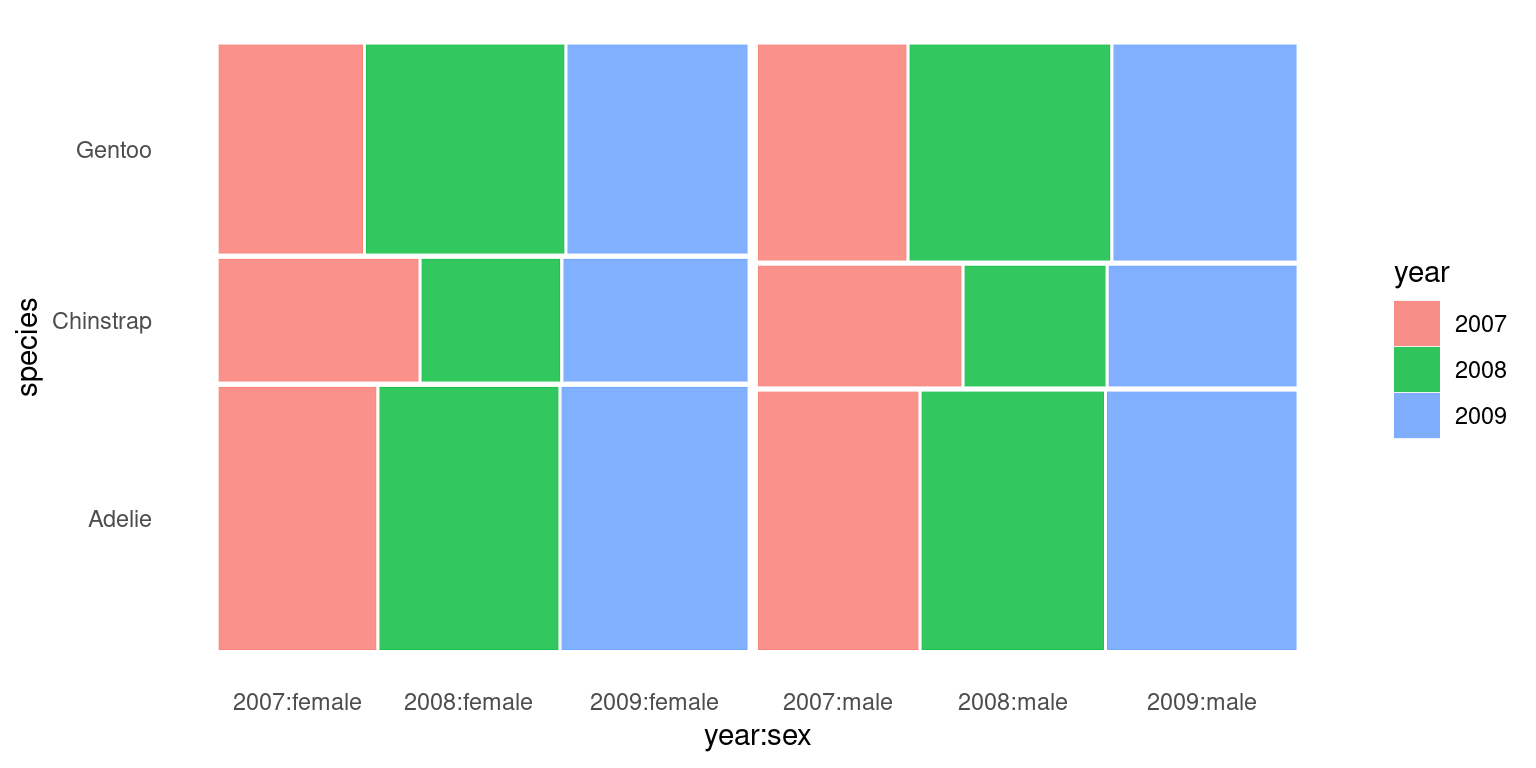
7.3.3 dot plot
Le dot plot est un nuage de point «dégénéré» au sens ou les cordonnées des points n’ont pas de signification autre que de les «localiser» dans une des modalités des variables qualitatives dont on représente la (table de) contingence.
ggplot(data = penguins %>% na.omit(), aes(y=species,x=sex))+
geom_jitter(width = 0.2, height = 0.2, cex=0.3, color= "darkcyan")+
theme_light()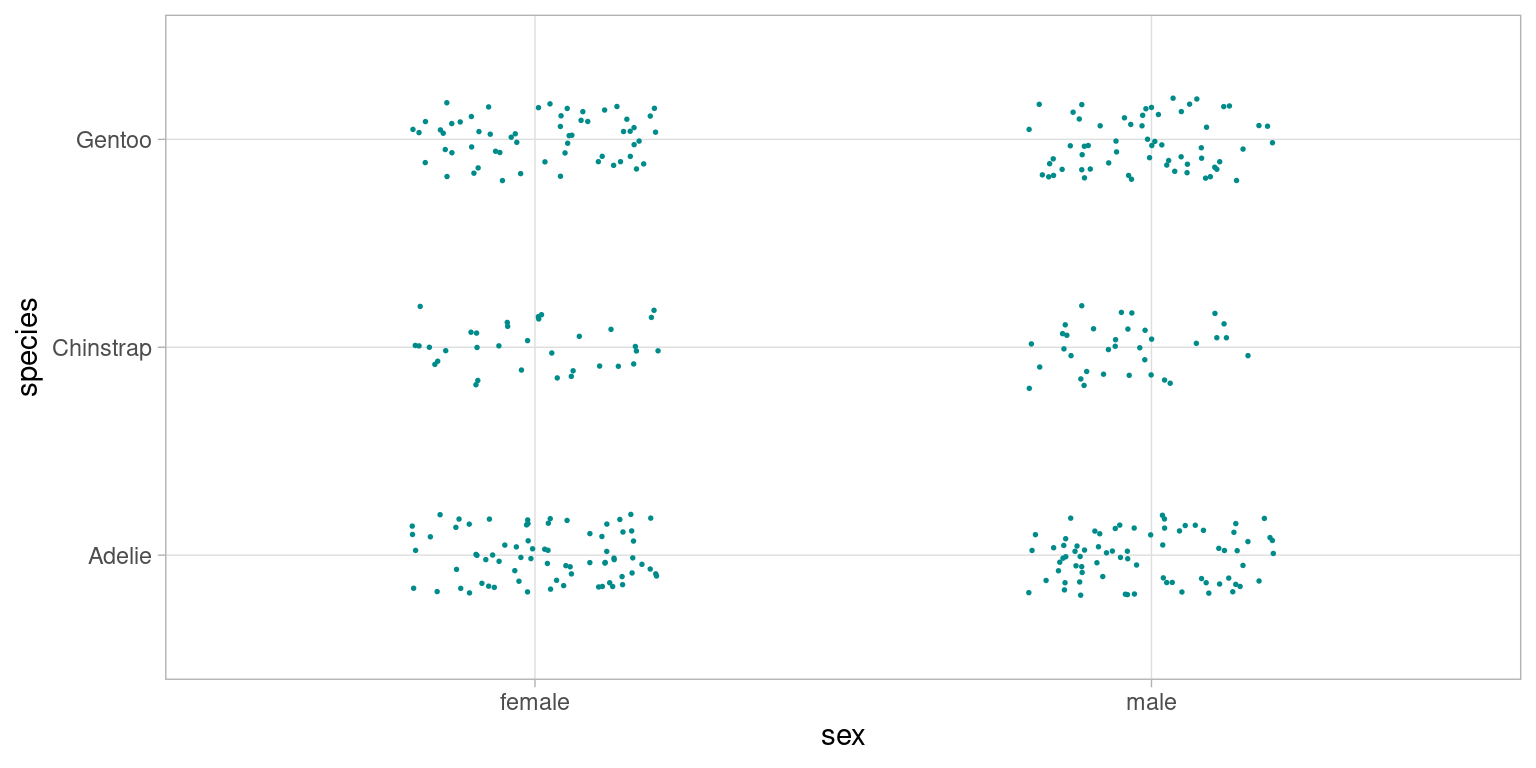
Cette représentation reste peu efficace : il est difficile de saisir d’un coup d’oeil les différences faibles d’effectif au croisement des modalités des variables, sauf en cas d’écarts importants.
Il est préférable d’afficher les effectifs des classes pour faciliter la lecture, comme le montre l’exemple ci-dessous.
Pour obtenir ces labels, il faut calculer “à part” la table de contingence ( object conting_sex_spec dans le code ci-dessous) en gardant les noms de colonnes inchangées, pour que ggplot puisque positionner les étiquettes (geom_label) aux bons endroits. Une troisième variable peut être utilisée éventuellement pour la couleur des points, ici la variable island
sex_spec_no_NA <- penguins %>% na.omit()%>% select(species,sex) # table de contingence
conting_sex_spec <- table(sex_spec_no_NA) %>% as.data.frame()
head(conting_sex_spec)# aperçu de la table de contingence## species sex Freq
## 1 Adelie female 73
## 2 Chinstrap female 34
## 3 Gentoo female 58
## 4 Adelie male 73
## 5 Chinstrap male 34
## 6 Gentoo male 61ggplot(data = penguins %>% na.omit(), aes(y=species,x=sex))+
geom_jitter(width = 0.2, height = 0.2, cex=0.5, aes(color=island))+
geom_label(data = conting_sex_spec,aes(label=Freq), nudge_y= -0.4 )+
theme_light()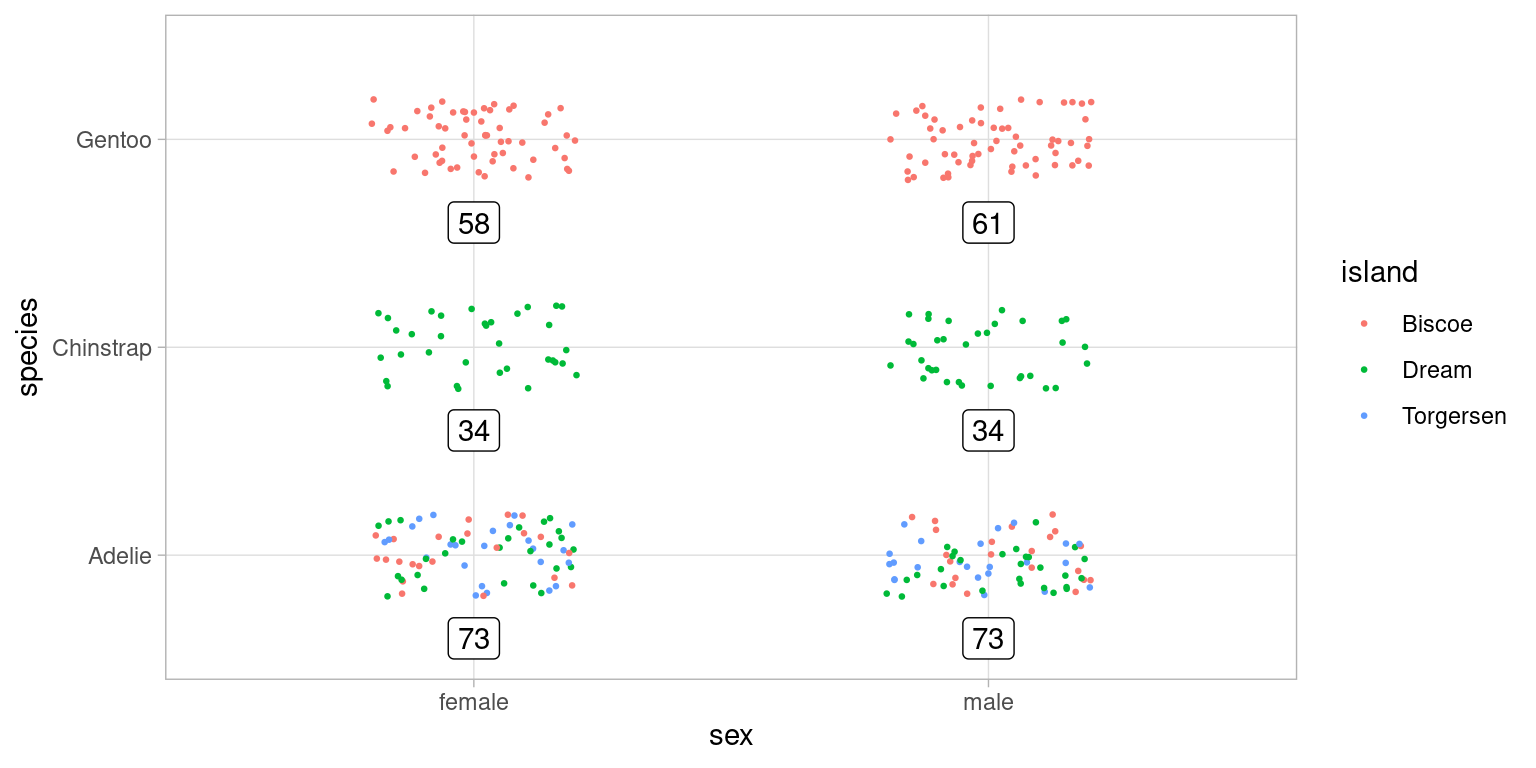
7.4 Lien entre plusieurs variables qualitatives
7.4.1 Flux sur axes paralllèles
Je ne sais pas trop comment traduire «parallel sets», le nom donné à ce genre de graphiques. On trouve parfois également le nom “alluvial graph”
En représentant les axes des variables qualitatives comme des axes parallèles gradués par leur modalités, et les effectifs comme un flux dont la taille est représentée par son épaisseur, on peut représenter plusieurs variables qualitatives les unes à côté des autres.
On utilise pour les créer le package ggforce.
Plusieurs étapes préparatoires sont nécessaires pour obtenir ce graphique :
- filtrer les valeurs NA
- ne conserver que les variables qualitatives
- obtenir la table de contingence. Celle ci est un tableau à \(n\) dimensions avec \(n\) le nombre de variables qualitatives retenues
- mettre à plat ce tableau à \(n\) dimension avec la fonction
meltdu packagereshape2 - transformer le format pour le rendre compatible avec le diagramme de flux du package
ggforce, à l’aide de la fonction dédiéegather_set_data
library(ggforce)
penguins_noNa <- penguins %>% na.omit() # filtrage valeurs NA
qualiVarspenguins <- penguins_noNa%>% select(c(species, island, sex)) # selection des variables qualitatives
table_conting_peng <- table(qualiVarspenguins) # table de contingence , tableau à 3 dimensions
table_conting_peng2 <- table_conting_peng %>% melt #rassemble et "aplatit" la table de contingence
flux_data <- gather_set_data(table_conting_peng2, 3:1) # mise au format pour le diagramme en flux
# constitution du graphique
ggplot(flux_data, aes(x, id = id, split = y, value = value)) +
geom_parallel_sets(aes(fill = sex), alpha = 0.5, axis.width = 0.1) +
geom_parallel_sets_axes(axis.width = 0.1) +
geom_parallel_sets_labels(colour = 'white')+
labs(title = "Espèce, île et sexe des manchots",
subtitle = "représentation en flux sur axes paralleles ")+
theme(panel.background = element_blank(),
panel.border = element_blank(),
panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
plot.background = element_blank(),
axis.ticks.y = element_blank(),
axis.ticks.x = element_blank(),
axis.title.x = element_blank(),
axis.text = element_blank()) 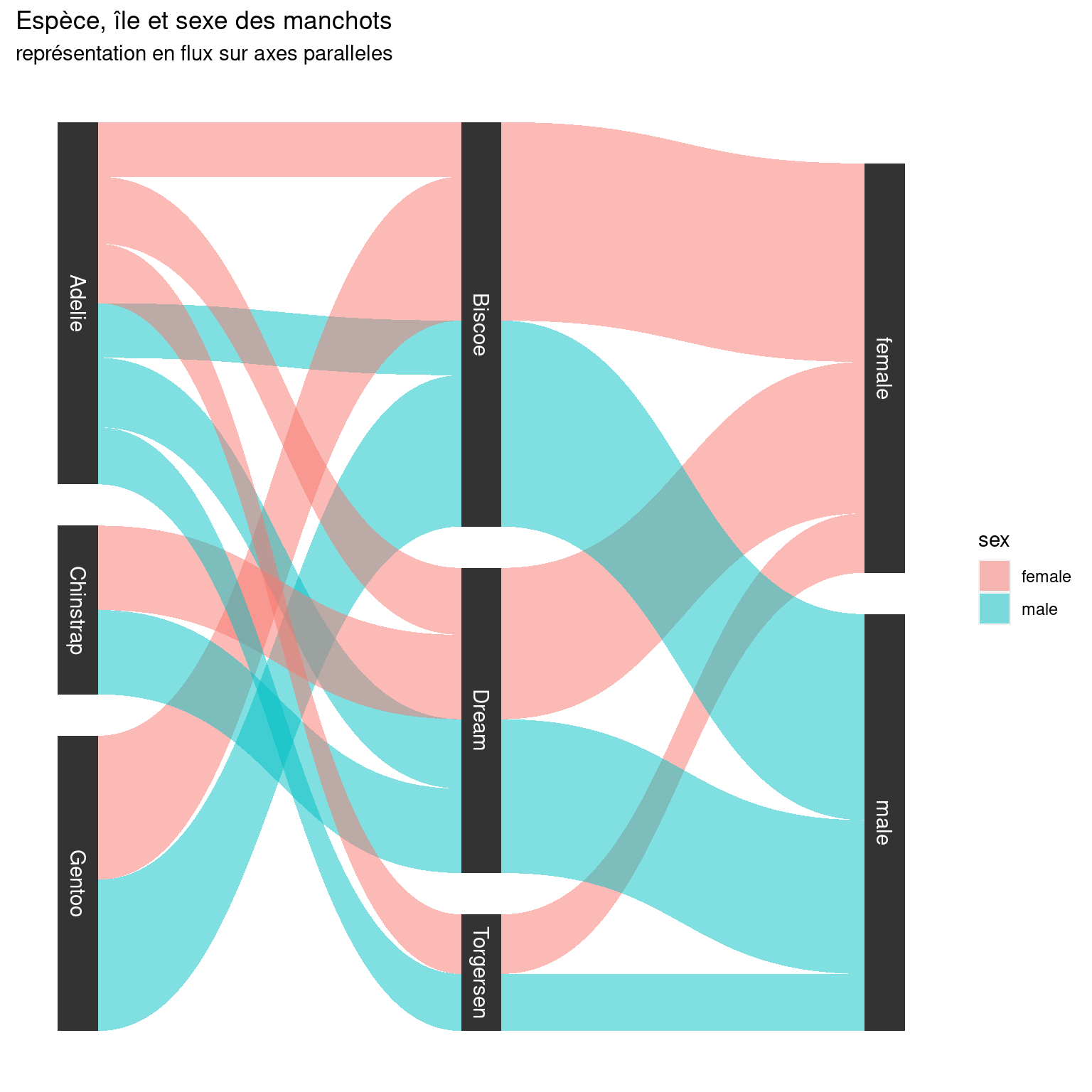
J’ai préféré retirer les axes et graduations , car celles-ci sont trompeuses, sur l’axe des \(y\) surtout, il est difficile d’interpréter l’épaisseur d’un flux car celle-ci varie dès qu’elle “sort” de sa variable. Cela dit, les proportions sont respectées, et sur cet exemple on voit bien que la proportions de mâles est toujours d’environ 50%, quelles que soient l’île et l’espèce.
7.5 Distributions de variables quantitatives
Les concepts derrière ces graphiques ont été abordés dans la section 2.1
7.5.1 Distribution
distribution <- ggplot(penguins, aes(x=body_mass_g))+
geom_density(color="grey12", fill="limegreen")+
labs(title = "Distribution de la masse des manchots")+
theme_light()
distribution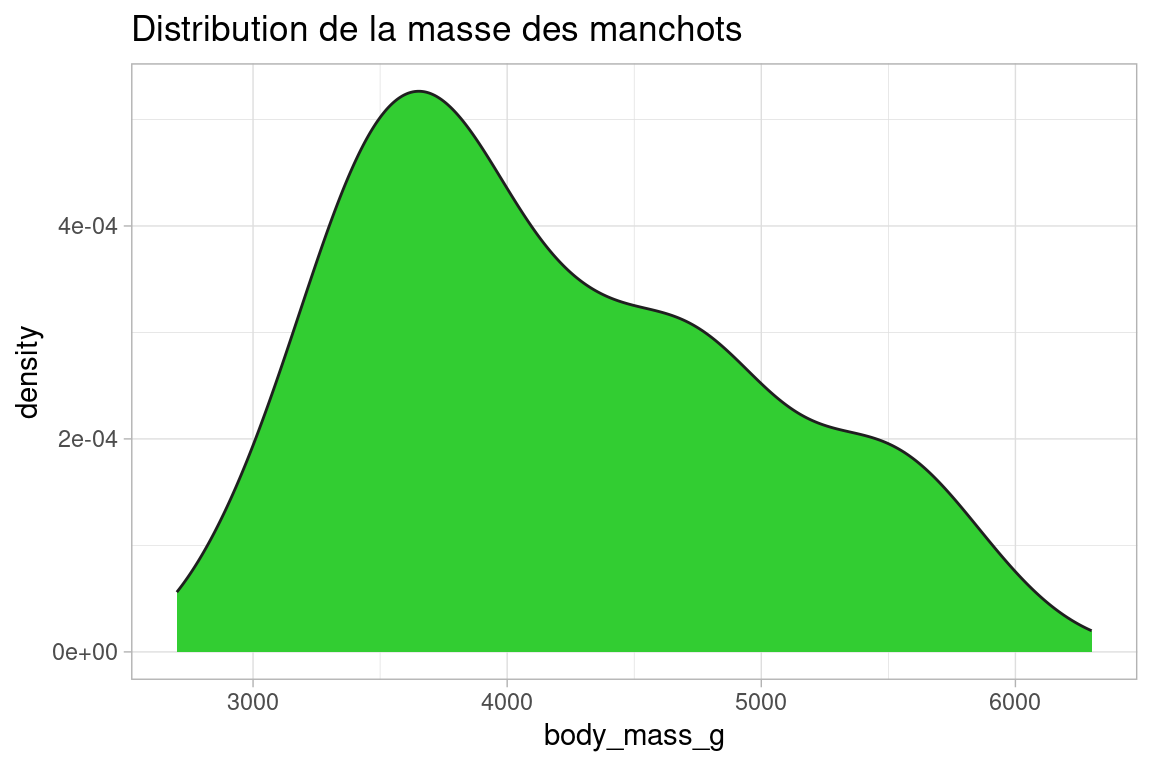
7.5.2 Histogramme
histogramme <- ggplot(penguins, aes(x=body_mass_g))+
geom_histogram(color="grey12", fill="limegreen", bins=25)+
labs(title = "Histogramme de la masse des manchots")+
theme_light()
histogramme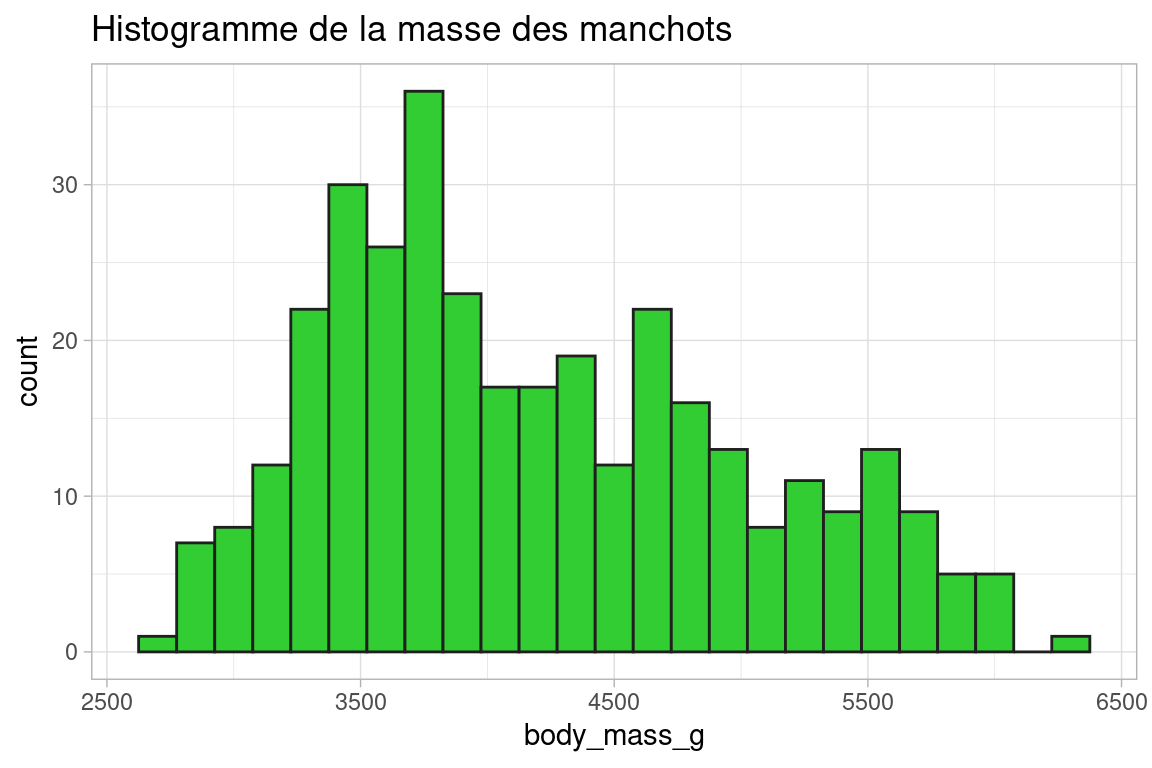
7.5.3 Distribution et histogramme
distrib_histo <- ggplot(penguins, aes(x=body_mass_g))+
geom_histogram(aes(y=..density..),color="grey80", fill="royalblue",)+
geom_density(color="grey12", fill="darkorange1", alpha= 0.4)+
labs(title = "Distribution de la masse des manchots", subtitle = "Histogramme et densité")+
theme_light()
distrib_histo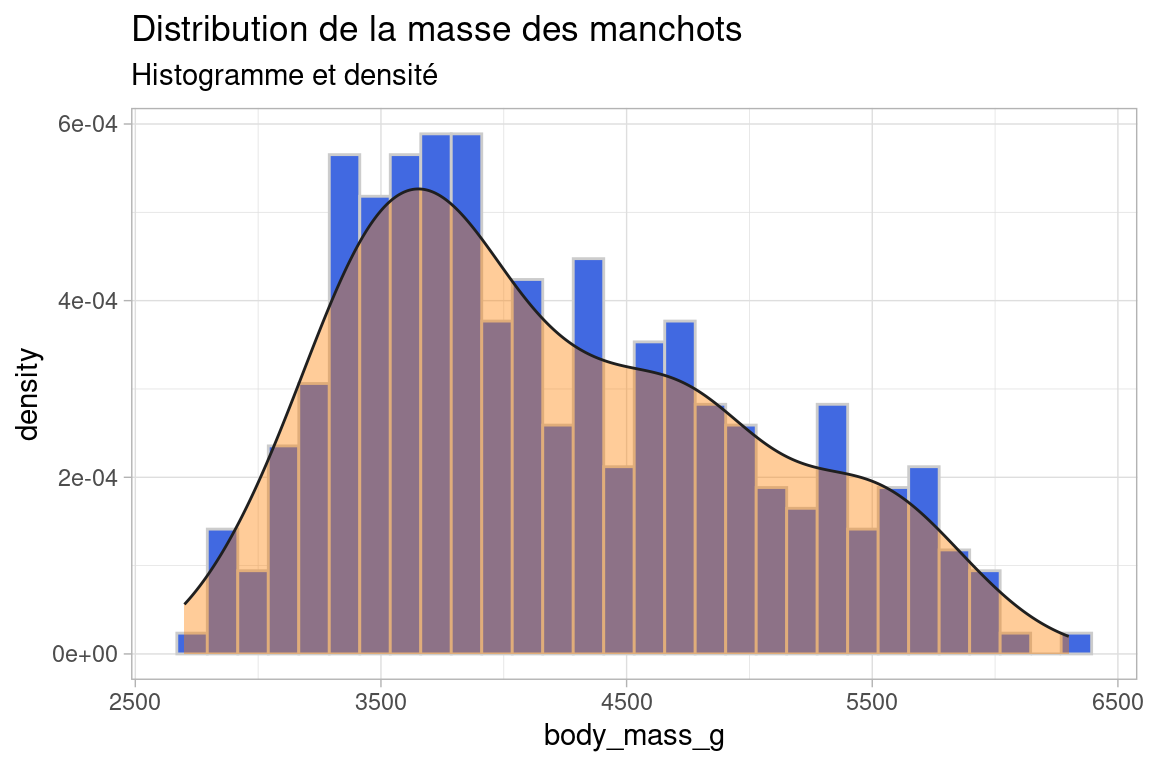
7.5.4 Pyramides (e.g. des âges)
Le graphique en pyramide est utile lorsque la population étudiée est décrite par une variable qualitative à deux modalités (binaire), \(V_1\) typiquement le sexe de nos manchots, et que nous voulons comparer la distribution d’une variable quantitative \(V_2\) dans les deux groupes formés par \(V_1\).
Obtenir une telle pyramide n’est pas immédiat, il faut ruser.
On commence par récupérer les données d’un histogramme :
- les graduations des centre des barres de l’histogramme (attribut mids de l’objet histogramme)
- les valeurs de rupture des classes
- les nombre d’individus (attributs counts) de chaque barre d’histogramme , pour chaque modalité de la variable qualitative binaire .
penguins_mass <- penguins %>% na.omit() %>% select(c(sex,body_mass_g)) #selection des variables
nb_class <- 10 # nombre de barre de l'histogramme
graduations <- hist(penguins_mass$body_mass_g,plot = F,breaks = nb_class)$mids # centres d'un histogramme à 10 classes
global_breaks <- hist(penguins_mass$body_mass_g,plot = F,breaks = nb_class)$breaks # rupture des classes de l'histogrammeCes classes étant définies pour toute la populations, on va compter le nombre de manchots mâles et femelles dans chacune d’elles, en recalculant un histogramme, mais cette fois pour chacune des sous populations avec les ruptures (global_breaks) définies à l’instant. :
#population male et femelle
penguins_mass_M <- penguins_mass %>% filter(sex=='male')
penguins_mass_F <- penguins_mass %>% filter(sex=='female')
counts_M <- hist(penguins_mass_M$body_mass_g,plot = F,breaks = global_breaks)$counts
counts_F <- hist(penguins_mass_F$body_mass_g,plot = F,breaks = global_breaks)$countsOn rassemble ensuite ces comptages dans un dataframe pour chacune des sous-populations.
Un ligne comporte une classe de mass (mass) , qui est en fait la valeur médiane des colonnes de l’histogramme global, et un nombre d’individus dans cette classe (population).
La population des mâles est rendu négative , de façon à l’afficher plus tard de l’autre coté de l’histogramme des femelles, en miroir.
On rassemble les deux sous-populations avec la fonction rbind.
df_histo_M <- data.frame(population= - 1 * counts_M, mass= graduations, sex="male") # NB : on prend les valeurs en négatif
df_histo_F <- data.frame(population= counts_F, mass= graduations, sex="female")
df_histo_MF <- rbind(df_histo_F,df_histo_M)Nos données sont prètes, on va maintenant représenter ces colonnes avec la géométrie geom_col de ggplot2, qui est une geométrie proche de celle de l’histogramme, mais plus flexible, puisqu’on on définit la position d’une colonne avec la variable x mais également la hauteur de chaque barre avec la variable visuelle y
pyramide <- ggplot(df_histo_MF , aes(x=mass , y=population)) +
geom_col(aes(fill=sex), color="white")+
theme_light()
pyramide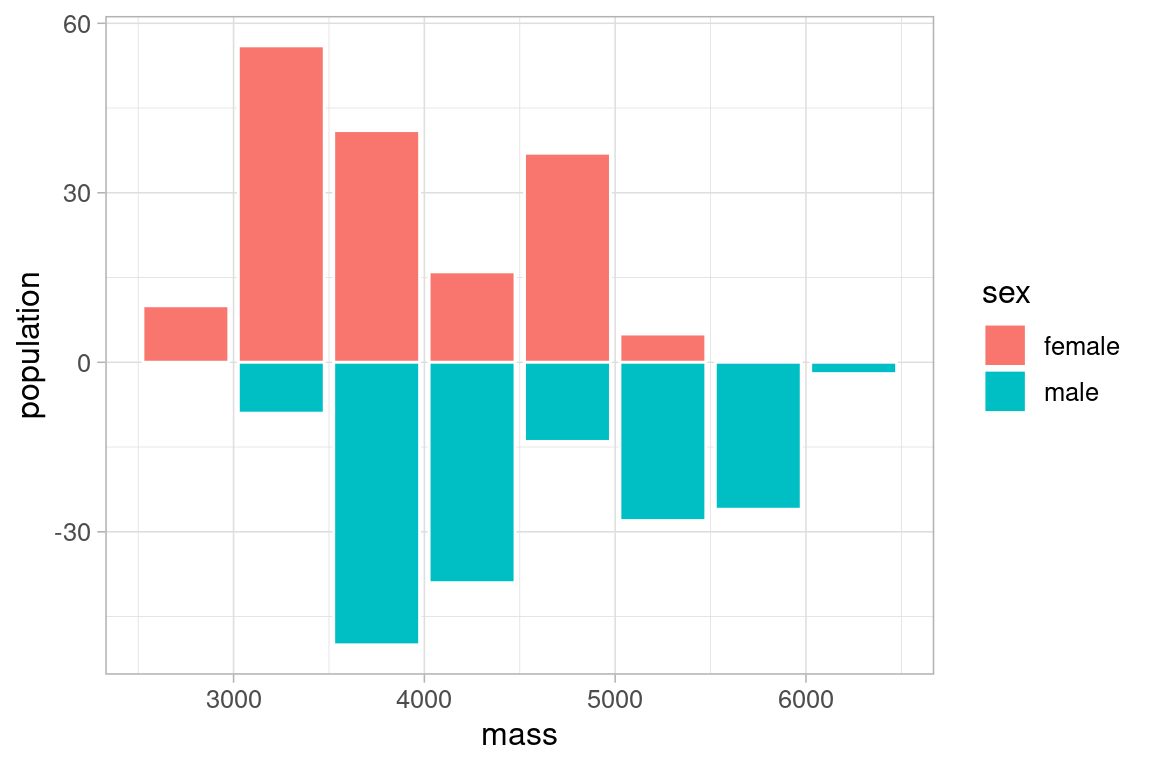
Il nous reste à maquiller l’axe des \(y\) puis à tourner le graphique.
Les valeurs des graduations, pour toutes les classes masses , y compris celles des ma^les qui sont négatives, sont obtenur à partir
de l’étendue (i.e. le segment \([min, max]\)) avec la fonction range(), qu’on va découper en quelques graduations régulières avec la fonction pretty , qui choisit des valeurs “rondes”.
Dans un deuxième temps, pour maquiller les étiquettes on prend la valeur absolue de ces graduations régulières.
On tourne le graphique avec la fonction coord_flip
graduations_regulieres <- df_histo_MF$population %>% range() %>% pretty() # étendue puis découpage puis valeur absolue
labels_maquilles <- graduations_regulieres %>% abs()
pyramide <- ggplot(df_histo_MF , aes(x=mass , y=population)) +
geom_col(aes(fill=sex), color="white")+
theme_light()+
scale_y_continuous(breaks= graduations_regulieres, labels= labels_maquilles) +
coord_flip()
pyramide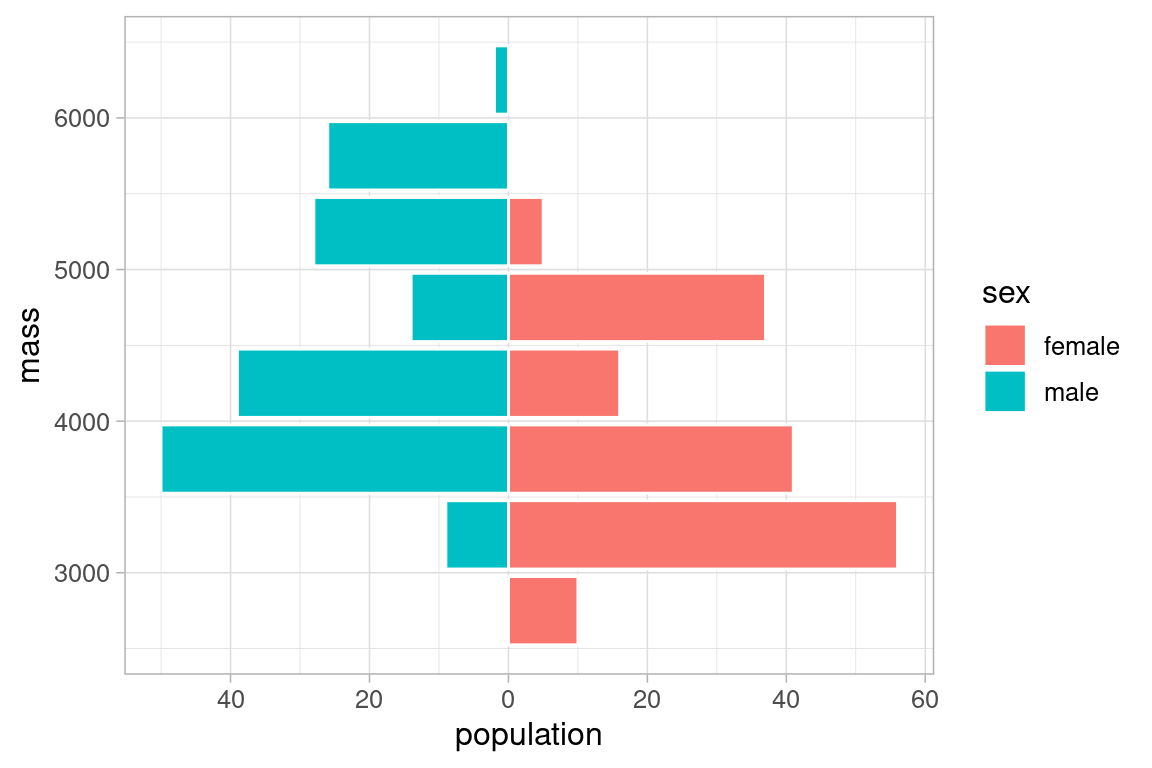
7.6 Distributions de variables qualitatives
Parler de distribution est un peu impropre, on pourrait utiliser le terme “répartition”
7.6.1 Bar chart
bar_chart <- ggplot(penguins, aes(x=species))+
geom_bar(color="grey80", fill="chartreuse4",)+
labs(title = "«Distribution» des espèces des manchots")+
theme_light()
bar_chart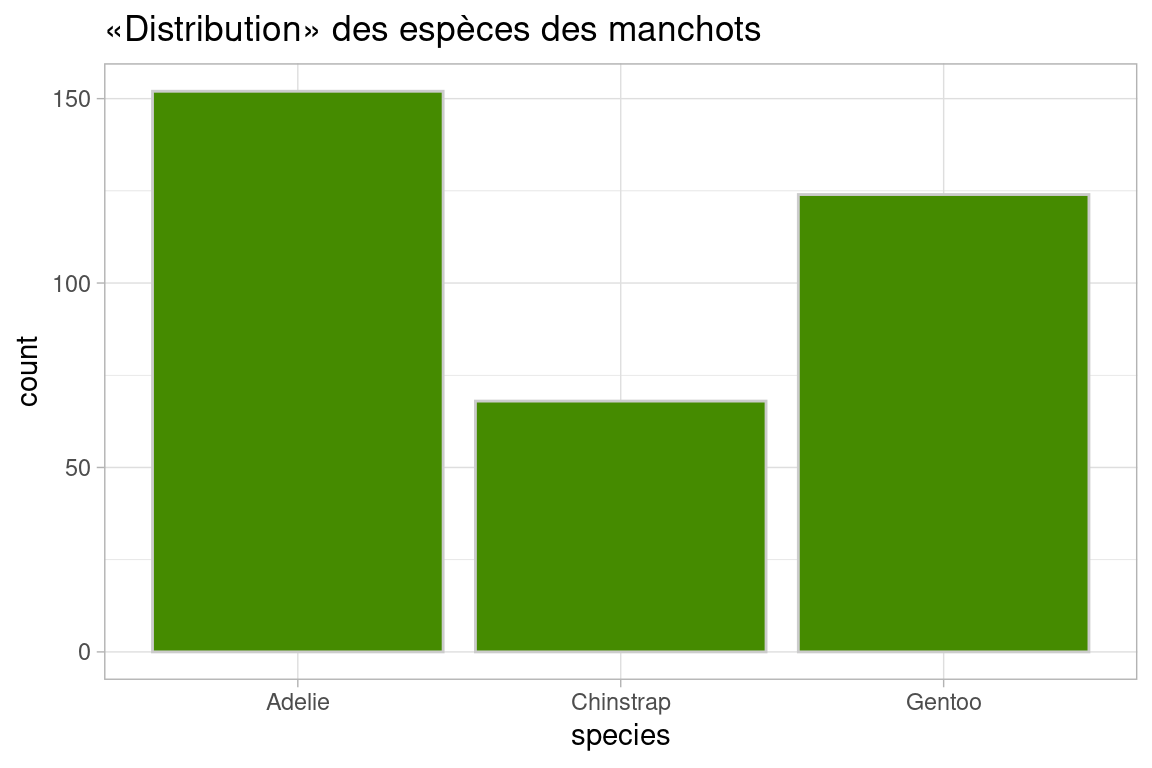
On eut également colorer les colonnes par la variable représentée :
bar_chart <- ggplot(penguins, aes(x=species))+
geom_bar(aes(fill=species), color="gray50", width= 0.5)+
labs(title = "«Distribution» des espèces des manchots")+
scale_fill_viridis_d()+
theme_light()
bar_chart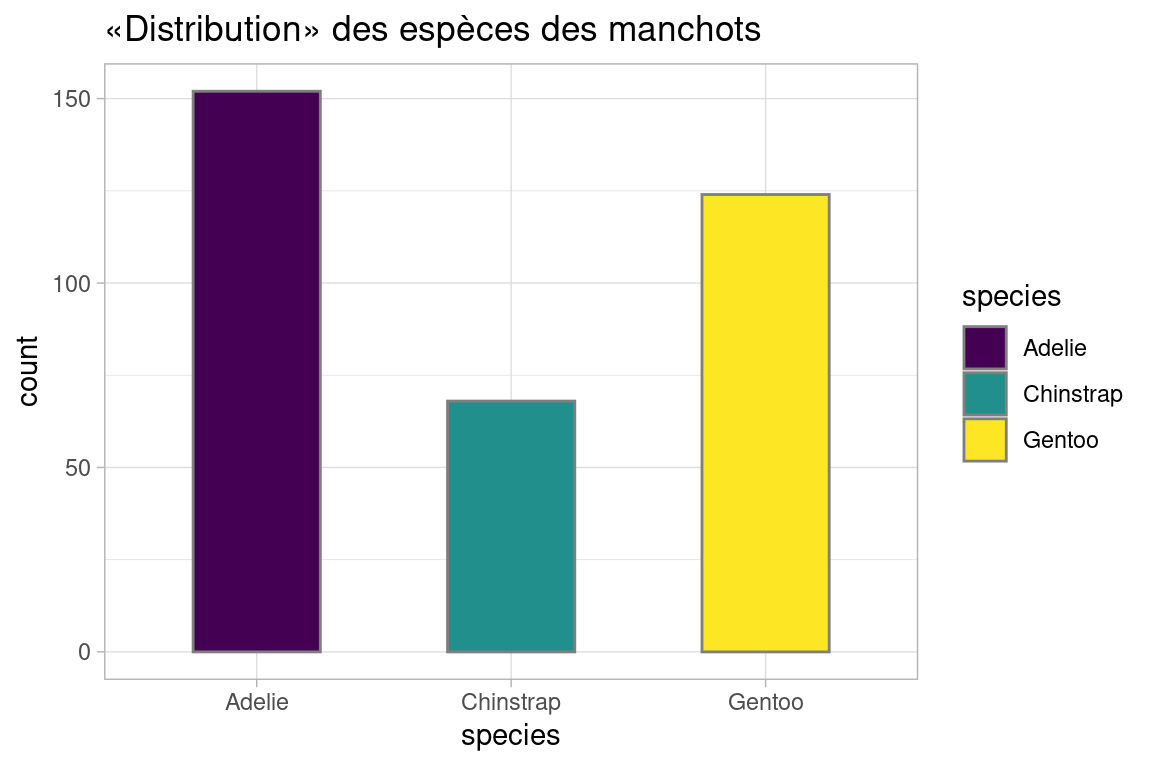
7.6.2 bar chart empilé
C’est une alternative préférable au diagramme en camembert. Quelques ajustement pour effacer les axes et leurs étiquettes sont nécessaires.
bar_chart <- ggplot(penguins, aes(y=""))+
geom_bar(aes(fill=species), orientation = "y")+
labs(title = "«Distribution» des espèces des manchots")+
ylab("")+
theme_light()
bar_chart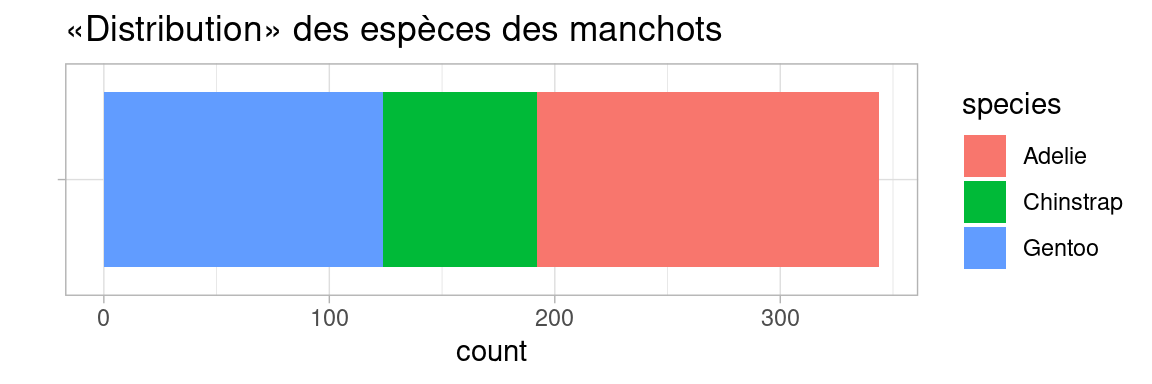
Ce type de graphes devient plus utile lorsqu’on veut représenter plusieurs répartitions à la fois :
bar_chart_2var <- ggplot(penguins, aes(x=island))+
geom_bar(aes(fill=species), orientation = "x", color="gray70", linewidth=0.2, width=0.4 )+
labs(title = "Espèces des manchots par île")+
scale_fill_viridis_d()+
theme_light()
bar_chart_2var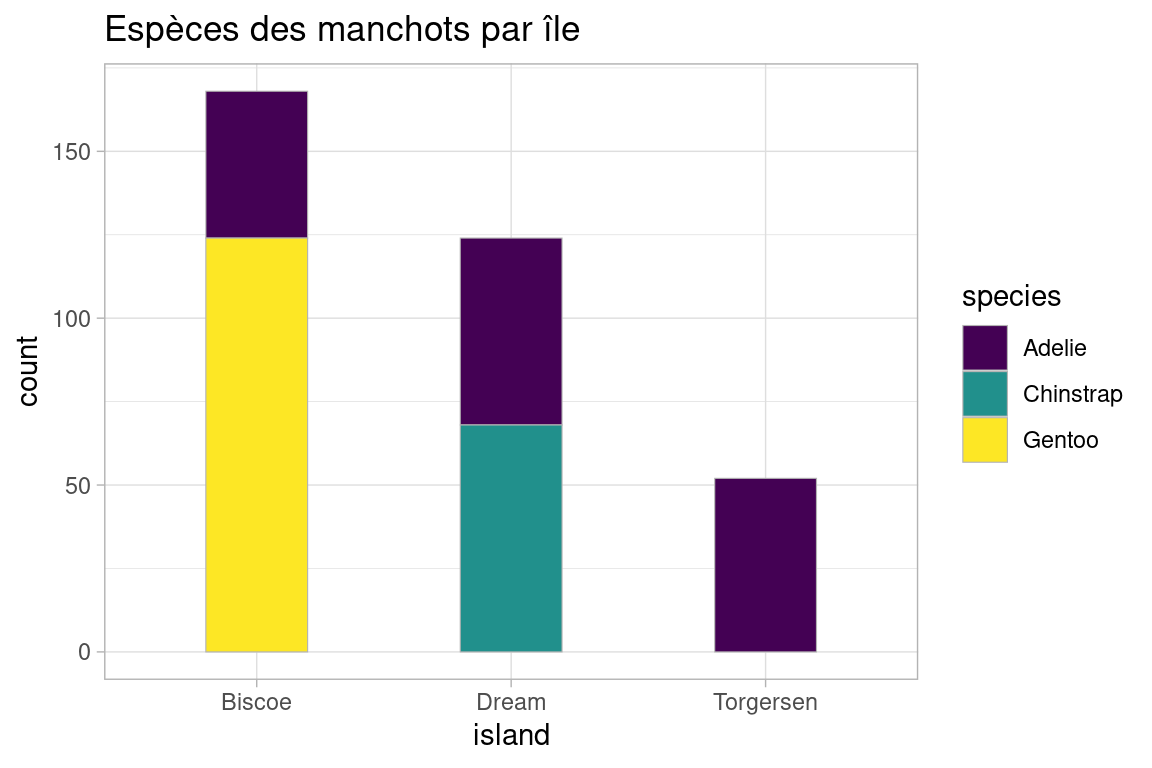
7.7 Graphiques “exotiques”
Les graphiques de cette section sont plus «impressionnants» mais pas forcément plus informatifs ni plus à même de transmettre de l’information sans biais.
7.7.1 Graphiques «en fleur»
Pour représenter de nombreuses répartitions, un bar chart classique prendrait trop de place en largeur ou en hauteur. on peut l’enrouler pour obtenir un cercle , plus compact :
On commence par représenter une variable, la masse, comme un bar chart, pour chaque manchot (on crée pour celà un ID) et on enlève les garnitures du graphes (labels et graduations de l’axe des X)
penguins_noNA <- penguins %>% na.omit() %>% sample_n(100)
penguins_noNA$ID <- 1:nrow(penguins_noNA)
flower <- ggplot(penguins_noNA, aes(x=ID, y= body_mass_g))+
geom_bar(aes(fill=species), orientation = "x", linewidth=0.1, stat="identity" )+
labs(title = "150 manchots")+
scale_fill_brewer()+
theme(axis.ticks.x = element_blank(),
axis.title.x = element_blank(),
axis.text.x = element_blank(),
panel.background = element_blank())
flower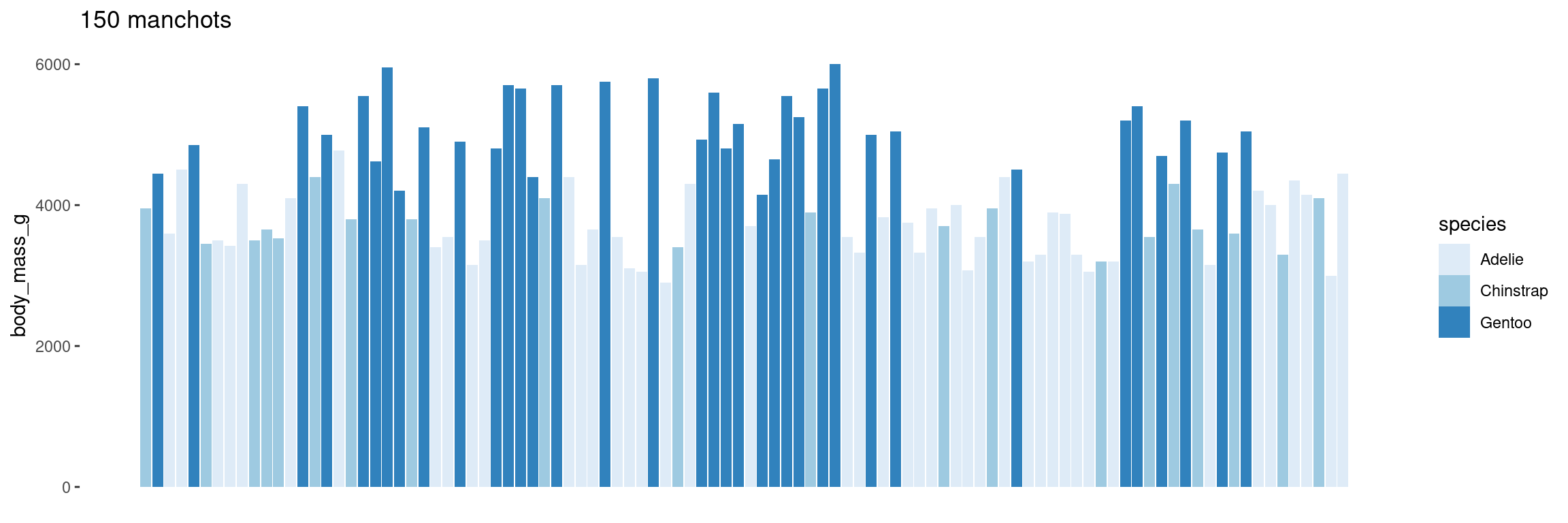 On transforme ensuite les coordonnées du graphes avec une transformation polaire , en on contrôle la taille du cercle avec
On transforme ensuite les coordonnées du graphes avec une transformation polaire , en on contrôle la taille du cercle avec ylim:
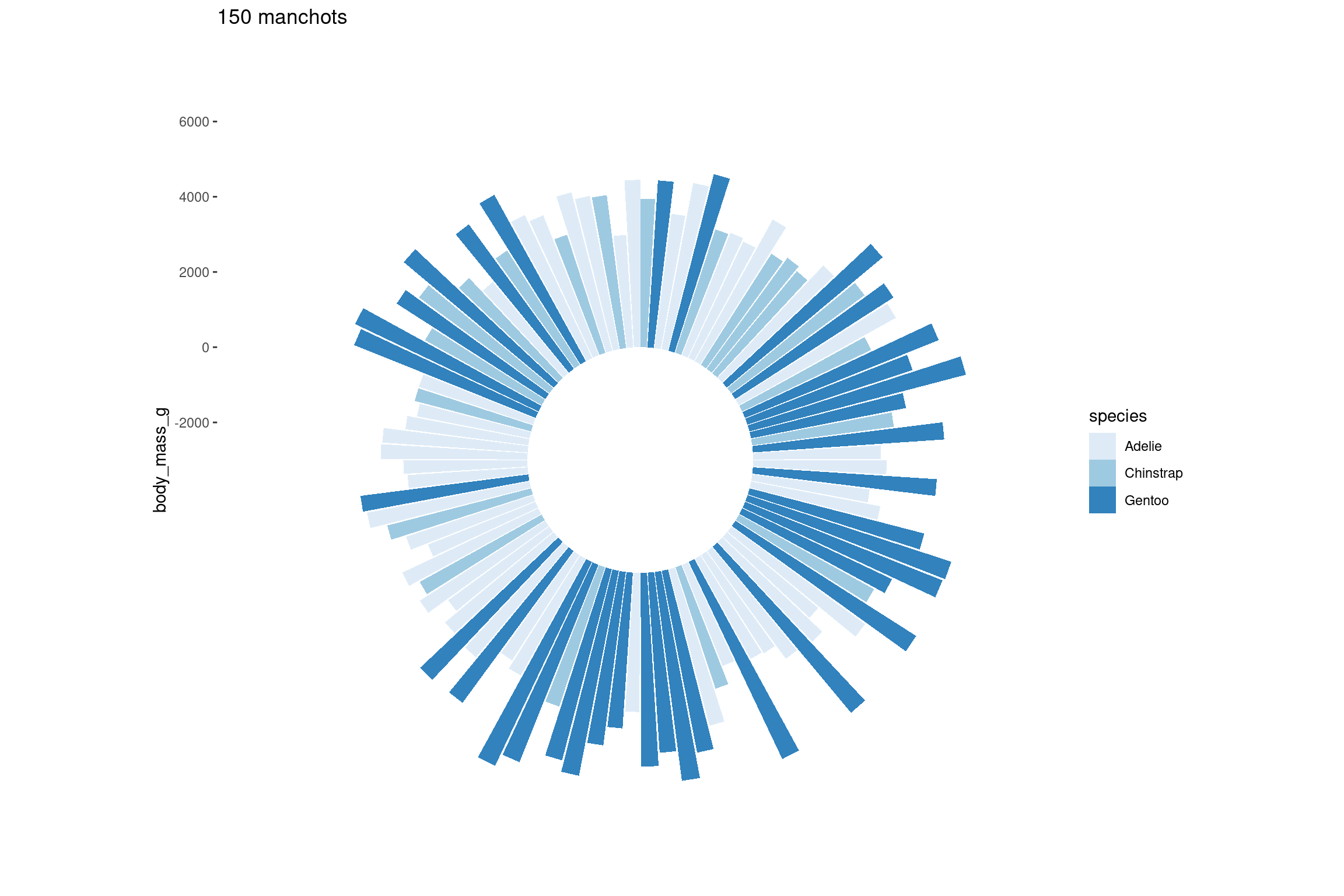
On peut également regroupper les individus, en triant les individus échantillonnés avant de recréer le graphe :
penguins_noNA <- penguins %>% na.omit() %>% sample_n(100) %>% arrange(species)
penguins_noNA$ID <- 1:nrow(penguins_noNA)
flower <- ggplot(penguins_noNA, aes(x=ID, y= body_mass_g))+
geom_bar(aes(fill=species), orientation = "x", linewidth=0.1, stat="identity" )+
labs(title = "Masse de 100 manchots de diverses espèces ")+
scale_fill_brewer()+
theme(axis.ticks.x = element_blank(),
axis.title.x = element_blank(),
axis.text.x = element_blank(),
panel.background = element_blank())
flower + coord_polar()+ ylim(-3000,6000) 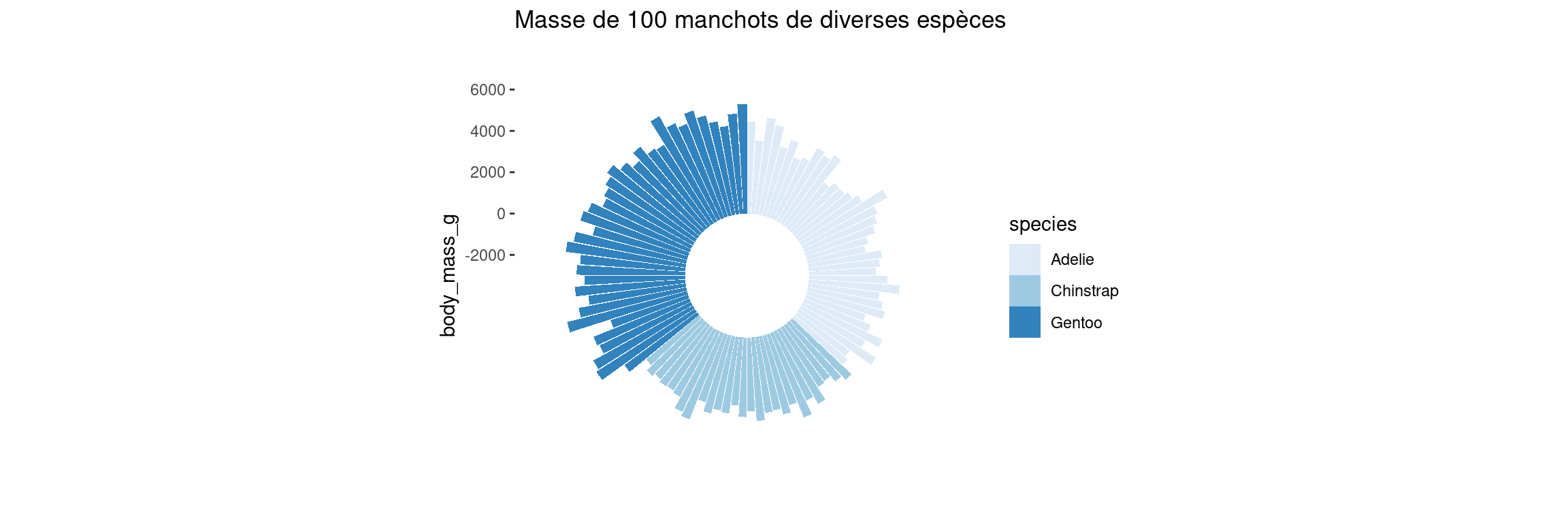
On remarque à quel point il est difficile de lire facilement les masses des individus. Seul le tri par espèce permet d’établir des relations : Adélie et Chinstrap sont à peu près de la même masse, les Gentoo sont plus lourds.