Chapitre 4 Visualiser une distribution avec R
Ce chapitre montre les différentes façons d’afficher une distribution, ainsi qu’un exemple de code R pour l’obtenir pour chacun des types de graphiques suivants .
- Histogramme
- Distribution
- BoxPlot
- Violin plot
- Pyramides (histogrammes juxtaposés)
- Polygones de fréquences
- Distribution cumulée , Fonction de répartition , CDF
- Dot strip plot
4.0.1 Histogramme : Code R + ggplot
library(palmerpenguins)
data(package = 'palmerpenguins')
mydata <- penguins
histo_mass <- ggplot(mydata)+
geom_histogram(aes(x=body_mass_g), fill="darkorchid4", color="darkgray", bins=50)+
labs(title = "Penguins Body Mass", subtitle = "Histogram")+
ylab("Count")+theme_light()
histo_mass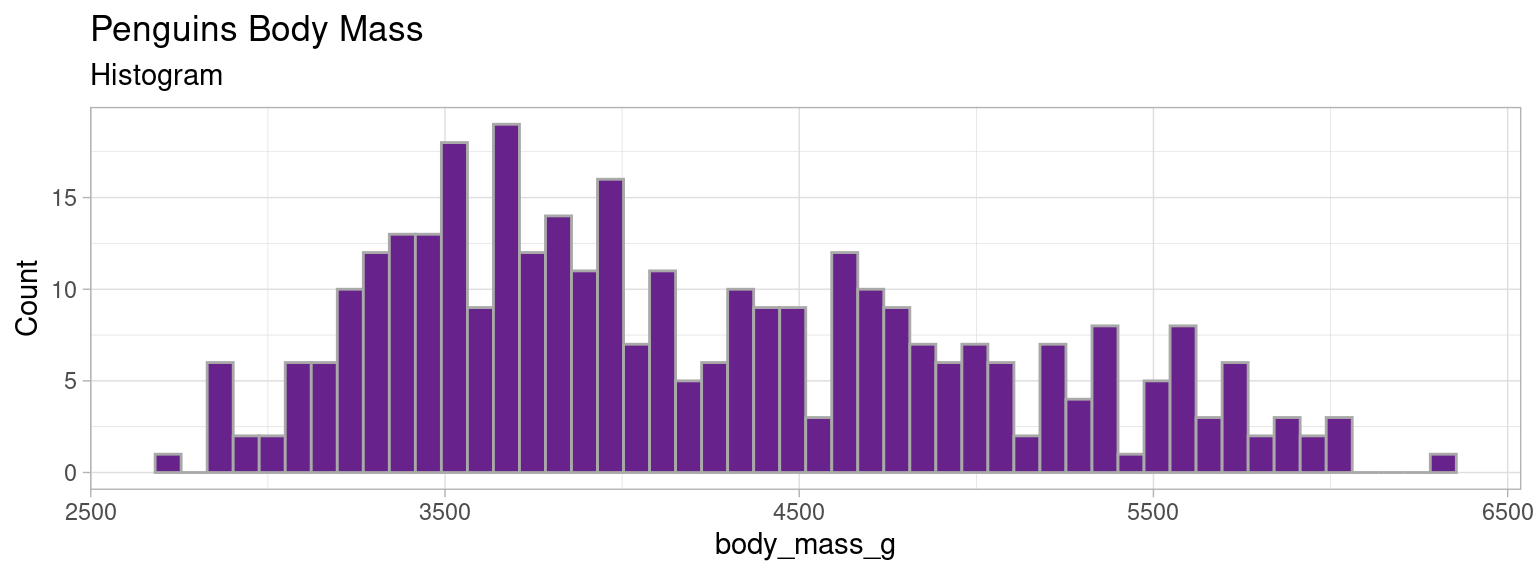
4.0.2 Distribution/densité : Code R + ggplot
⚠ ce n’est pas exactement une probabilité, mais une densité de probabilité. Pour obtenir la probabilité pour une valeur \(x\) , il faut intégrer sur un petit \(dx\) autour de \(x\) (cf. 2.1).
library(palmerpenguins)
mydata <- penguins
distrib_mass <- ggplot(mydata)+
geom_density(aes(x=body_mass_g), fill="darkorchid4", color="darkgray")+
geom_vline(aes(xintercept=mean(body_mass_g, na.rm = T)), color="black", size=0.4, linetype="dashed" )+
labs(title = "Penguins Body Mass", subtitle = "Probability density and mean")+
theme_light()
distrib_mass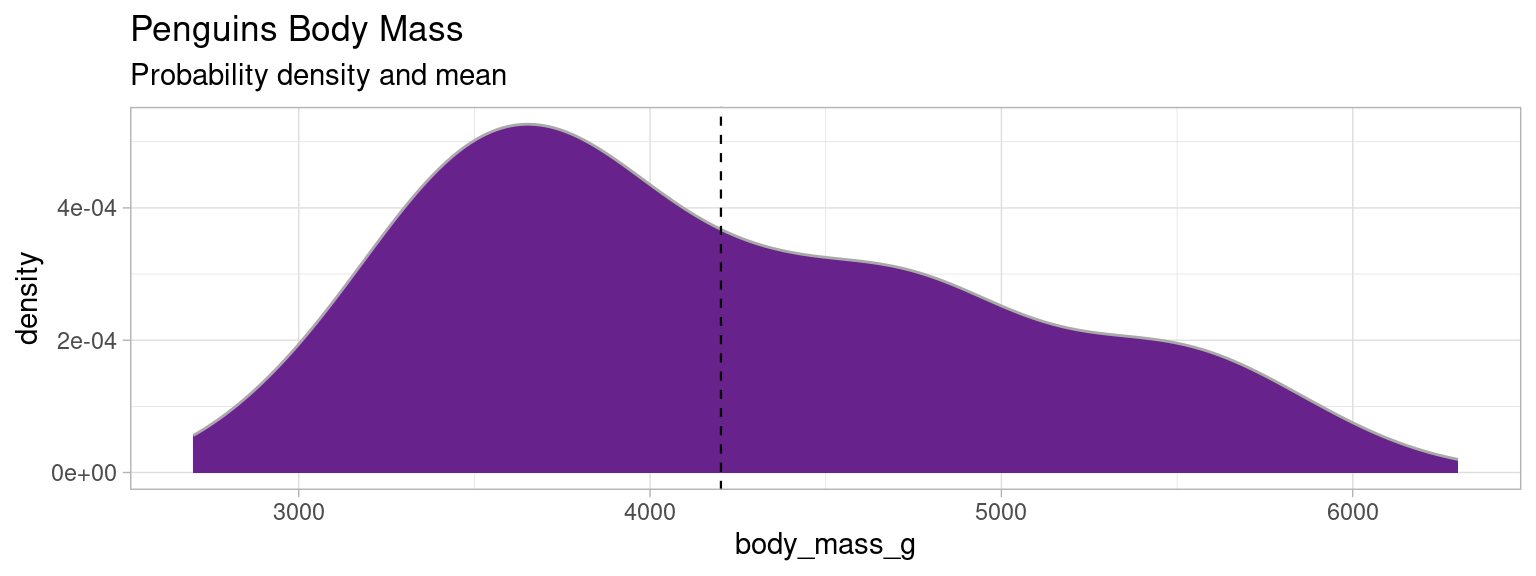
4.0.3 BoxPlot : Code R + ggplot
library(palmerpenguins)
mydata <- penguins
boxplot_mass <- ggplot(mydata)+
geom_boxplot(aes(x=body_mass_g, y=species, color=species))+
labs(title = "Penguins Body Mass", subtitle = "BoxPlot by Species")+
theme_light()
boxplot_mass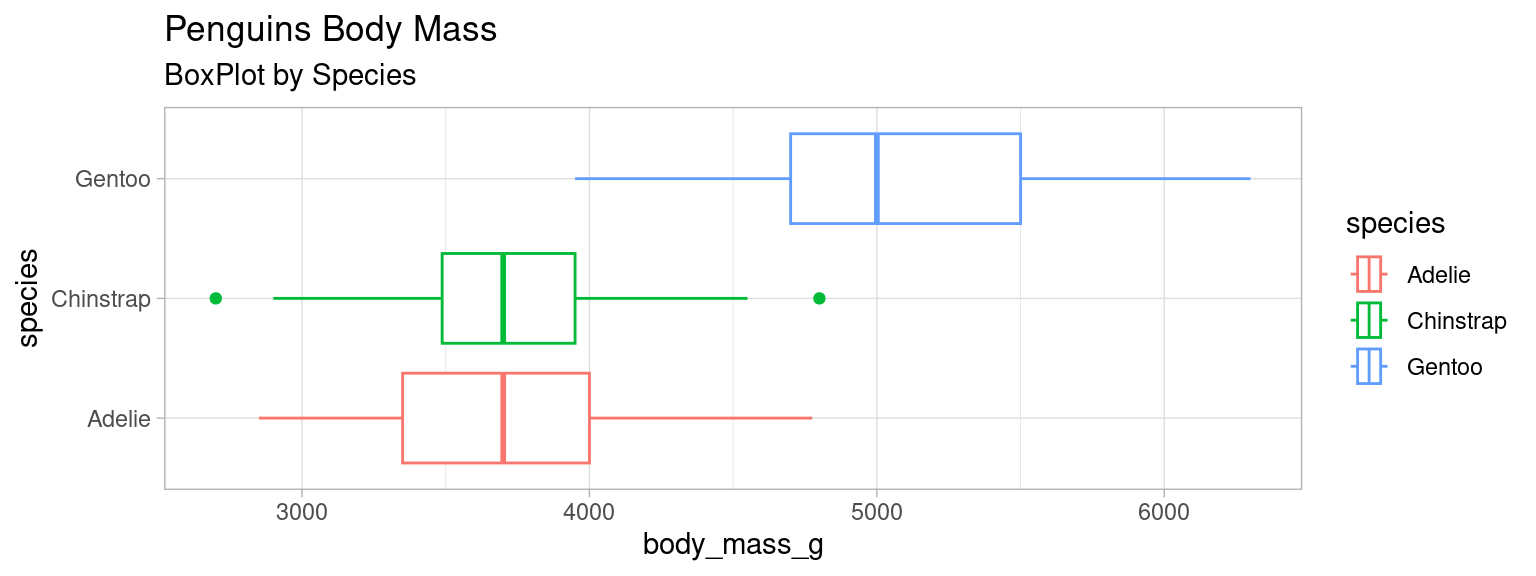
4.0.4 Violin plot : Code R + ggplot
Le violin plot est obtenu en faisant une symétrie de la distribution par rapport à l’axe des \(x\) d’un affichage classique de distribution.
Cette visualisation est utile pour des distributions complexes, e.g. mal résumées par la moyenne et la dispersion, observées pour plusieurs facteurs ou classe (comme ici, avec l’espèce des pingouins).
library(palmerpenguins)
mydata <- penguins
violin_mass <- ggplot(mydata)+
geom_violin(aes(y=body_mass_g, x=species, fill=species), color="gray", trim=F)+
labs(title = "Penguins Body Mass", subtitle = "ViolinPlot by Species")+
theme_light()
violin_mass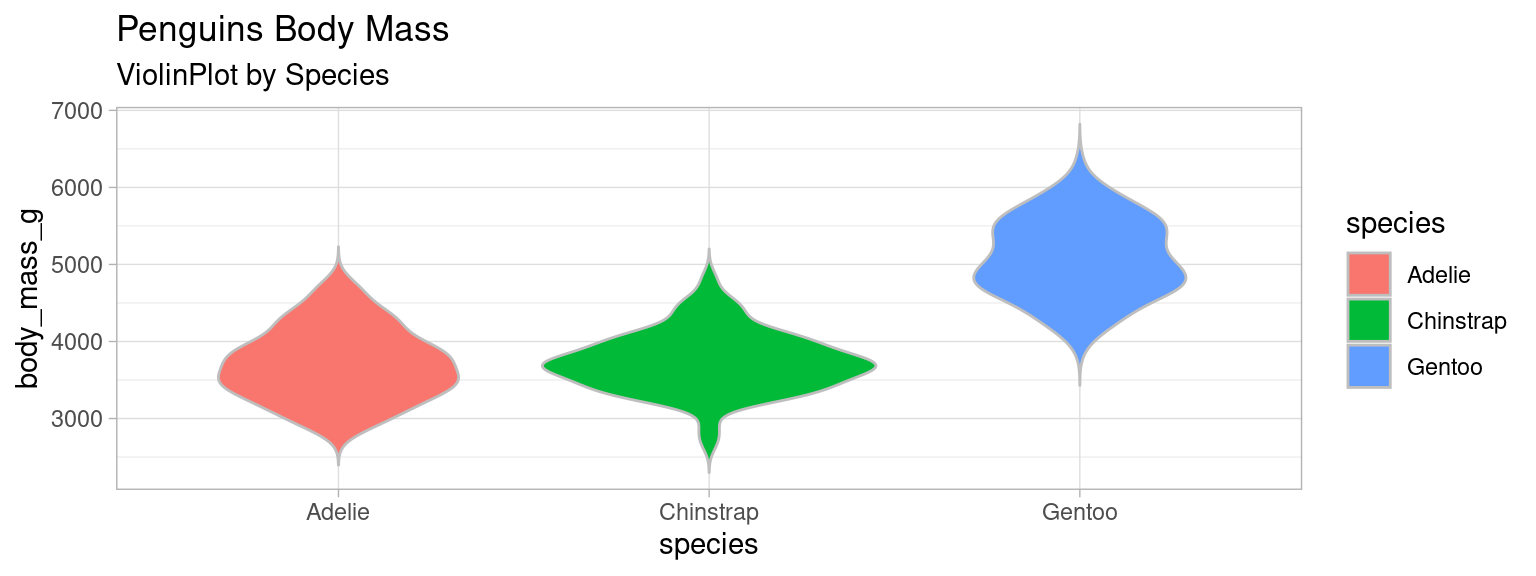
4.0.5 Violin plot et Boxplot : Code R + ggplot 2
On peut superposer un boxplot de la distribution d’une variable à son violin-plot.
library(palmerpenguins)
mydata <- penguins
violin_mass <- ggplot(mydata)+
geom_violin(aes(y=body_mass_g, x=species, fill=species), color="lightgray", trim=F)+
geom_boxplot(aes(y=body_mass_g, x=species, fill=species), color="black", fill="#eeeeee" ,width=0.1)+
labs(title = "Penguins Body Mass", subtitle = "ViolinPlot and Boxplot by Species")+
theme_light()
violin_mass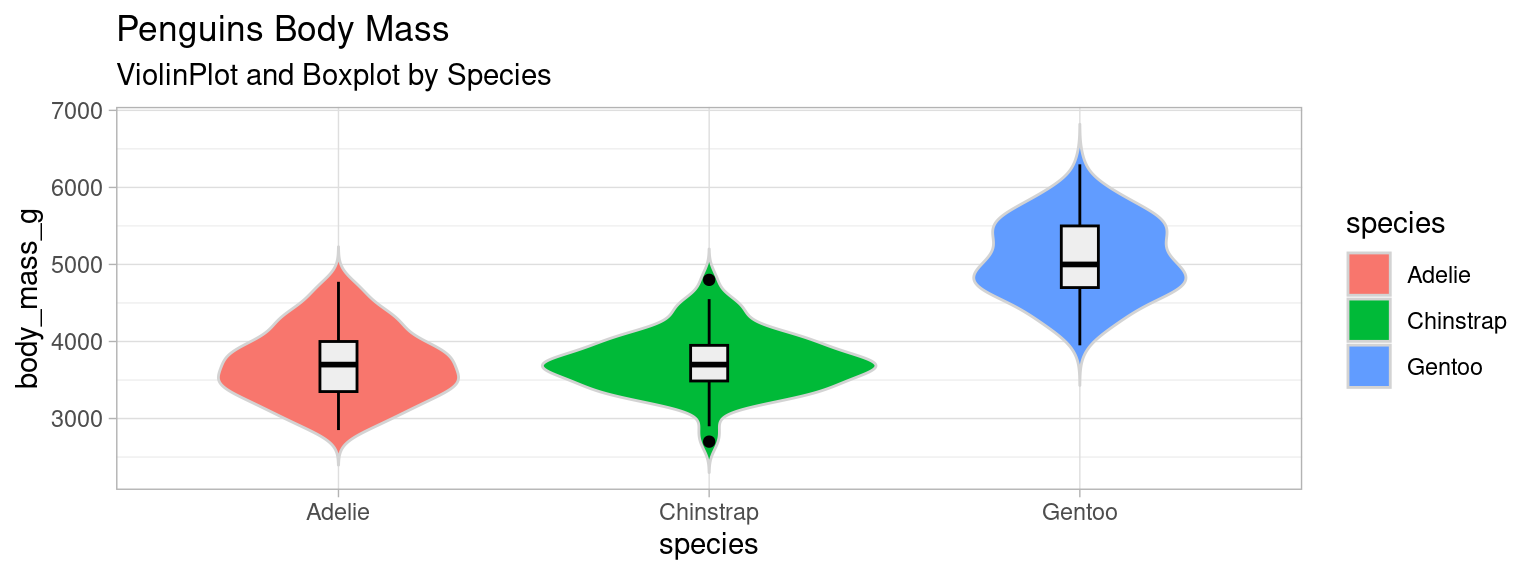
4.0.6 Pyramides (histogrammes juxtaposés)
Lorsqu’une des variables est qualitative à deux modalités, on peut classer les individus selon cette variable en deux sous-populations, et représenter les histogrammes d’une autre variable quantitative pour ces deux sous-populations :
library(palmerpenguins)
mydata <- penguins
pyramide_mass <- ggplot(mydata, aes(fill = sex)) +
geom_bar(data = subset(mydata, sex == "female"), stat = "bin", aes(x=body_mass_g, y=..count..*(-1)), color="grey") +
geom_bar(data = subset(mydata, sex == "male"), stat = "bin", aes(x=body_mass_g), color="grey") +
scale_y_continuous(labels = paste0(as.character(c(seq(20, 0, -10), seq(10, 20, 10))))) +
ylab("count")+ coord_flip()+
labs(title = "Penguins Body Mass", subtitle = "Pyramid Plot by sex")+
theme_light()
pyramide_mass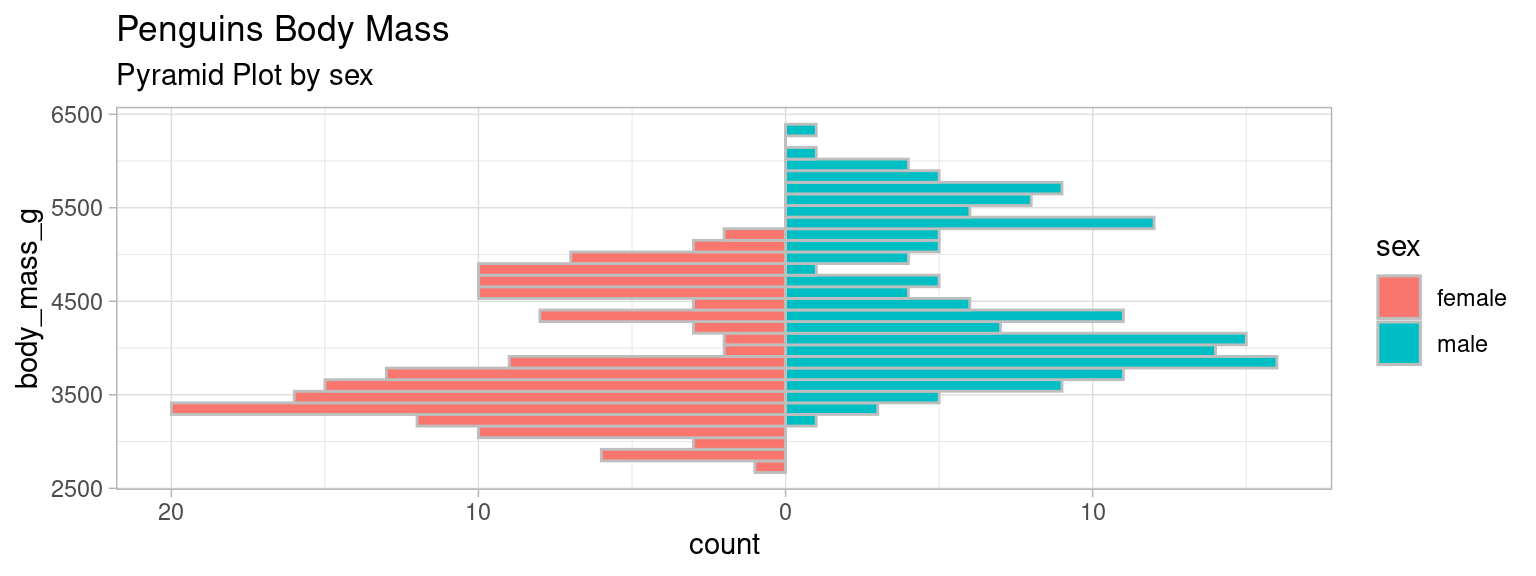
Une version très connue de cette visualisation est la pyramide des âges, utilisée en démographie.
4.0.7 Polygones de fréquences
On peut voir cette visualisation de distribution comme un «histogramme en courbe»
Elle est utile pour comparer plusieurs distributions.
library(palmerpenguins)
mydata <- penguins
freqpoly_mass <- ggplot(mydata)+
geom_freqpoly(aes(x=body_mass_g, color=species), bins=50)+
labs(title = "Penguins Body Mass", subtitle = "Frequence Polygons")+
ylab("Count")+theme_light()
freqpoly_mass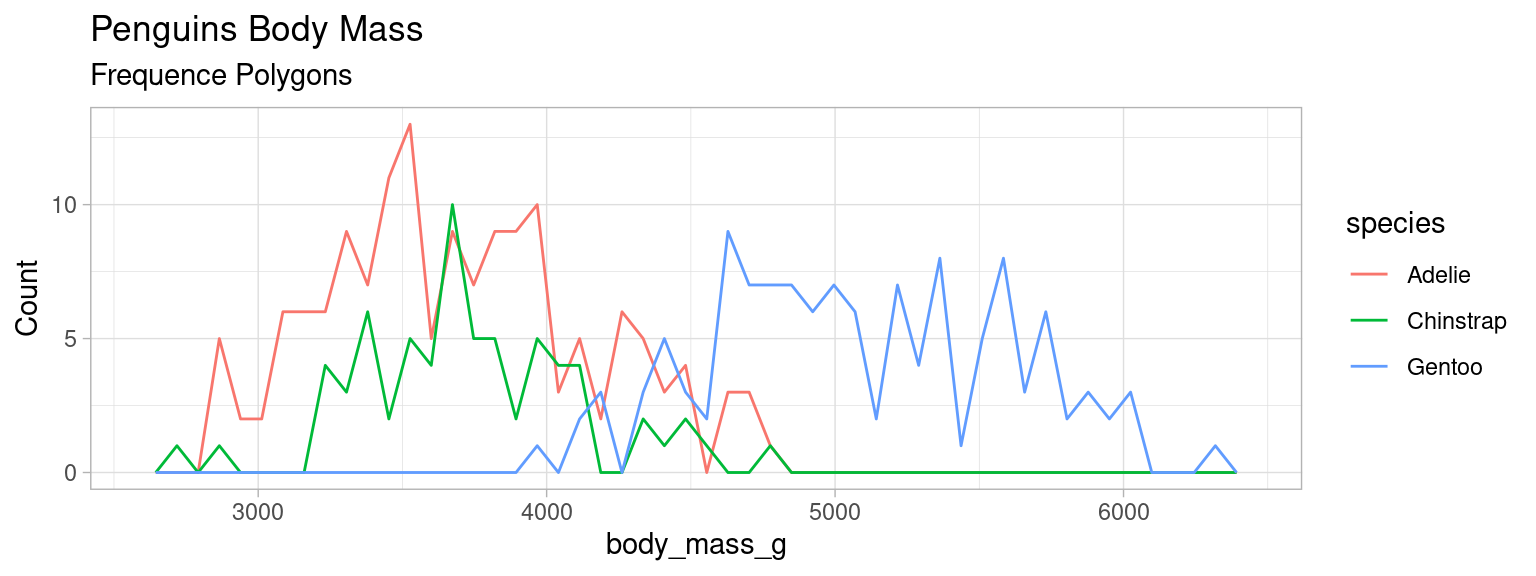
4.0.8 Distribution cumulée, Fonction de répartition, CDF
Les termes “Distribution cumulée”, “Fonction de répartition”, et “CDF” (Cumulative Distribution Function in english) sont en principe synonymes.
Cette représentation diffère des représentation habituelle des distributions car on représente sur l’axe des ordonnées non plus la valeur de l’effectif ou la densité, mais la somme cumulée de l’effectif ou de la densité.
Cette courbe indique en abscisse la valeur de la variable \(V\), et en ordonnée la probabilité empirique d’avoir dans la population, un individu pour lequel \(V\leq x\)
i.e. c’est la fonction
\[F_{V}(x)=\mathbb {P} (V\leq x)\]
library(palmerpenguins)
mydata <- penguins
plot_mass <- ggplot(mydata, aes(x= body_mass_g))+
stat_ecdf(color="darkorchid4")+
labs(title = "Penguins Body Mass", subtitle = "Cumulative Distribution Function")+
ylab("Probability")+theme_light()
plot_mass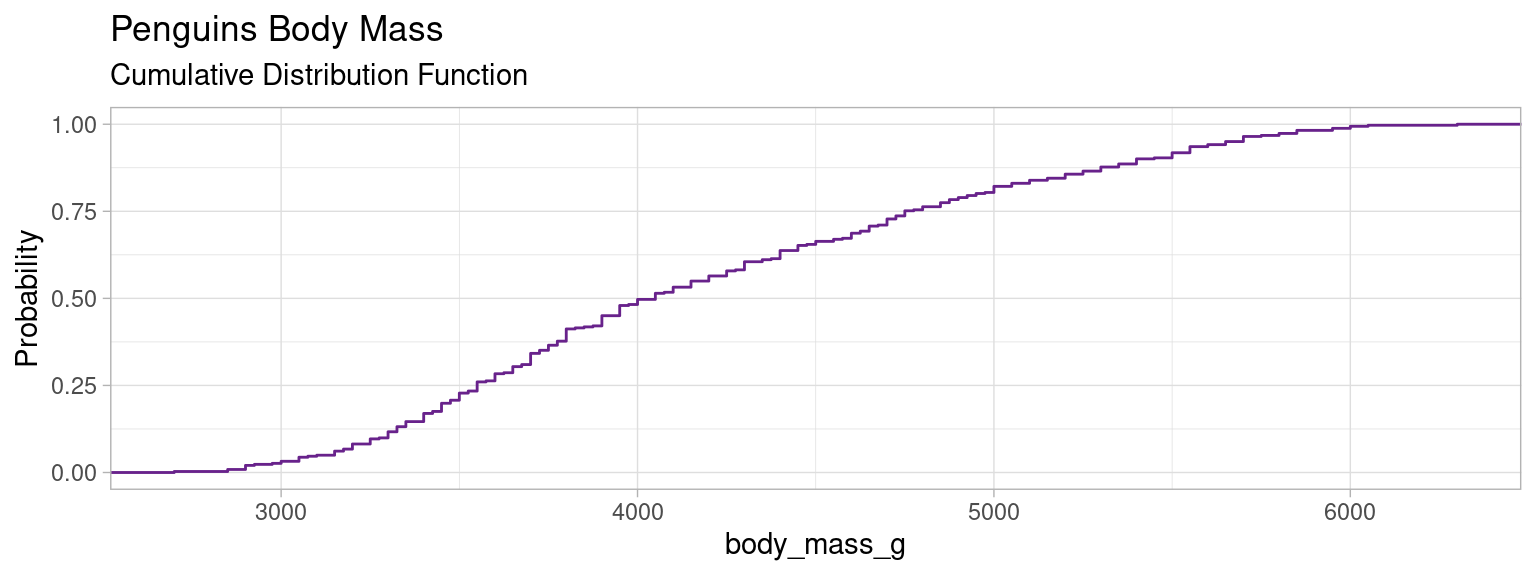
Cette représentation permet par exemple de superposer les CDF de sous groupes de la population (ici selon le facteur de l’espèce des penguins)
library(palmerpenguins)
mydata <- penguins
plot_mass <- ggplot(mydata)+
stat_ecdf(aes(x= body_mass_g, color=species))+
labs(title = "Penguins Body Mass by Species", subtitle = "Cumulative Distribution Function")+
ylab("Probability")+theme_light()
plot_mass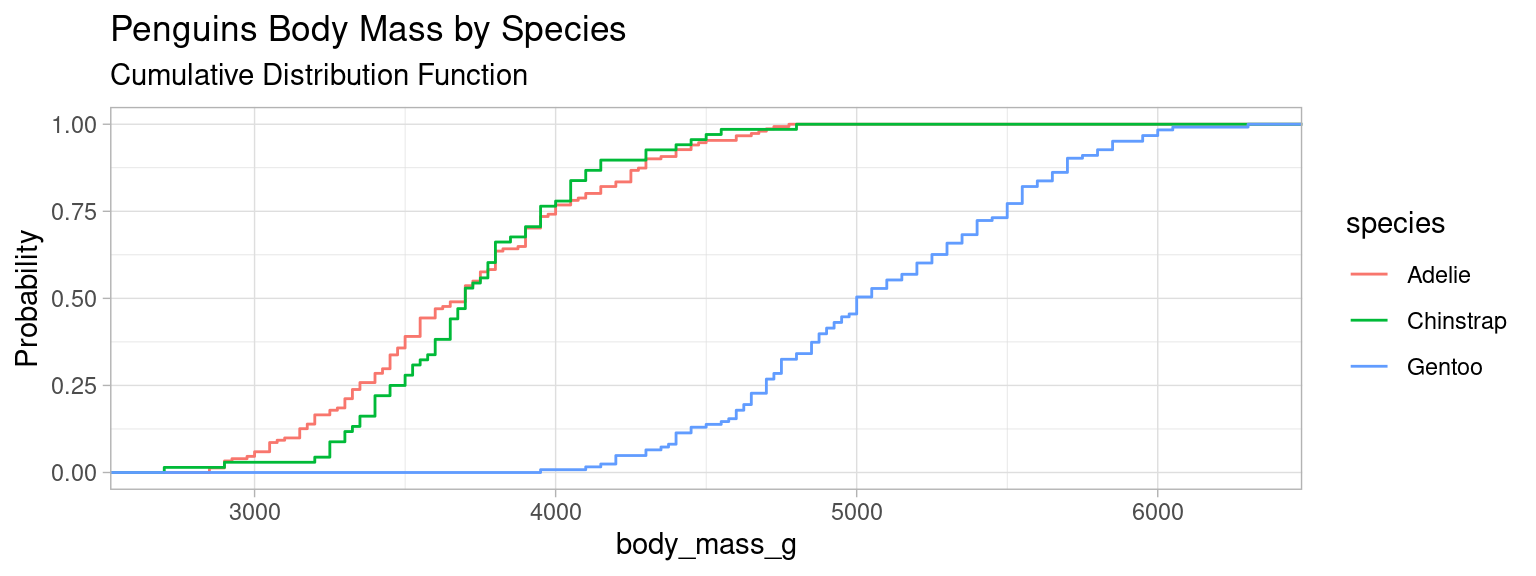
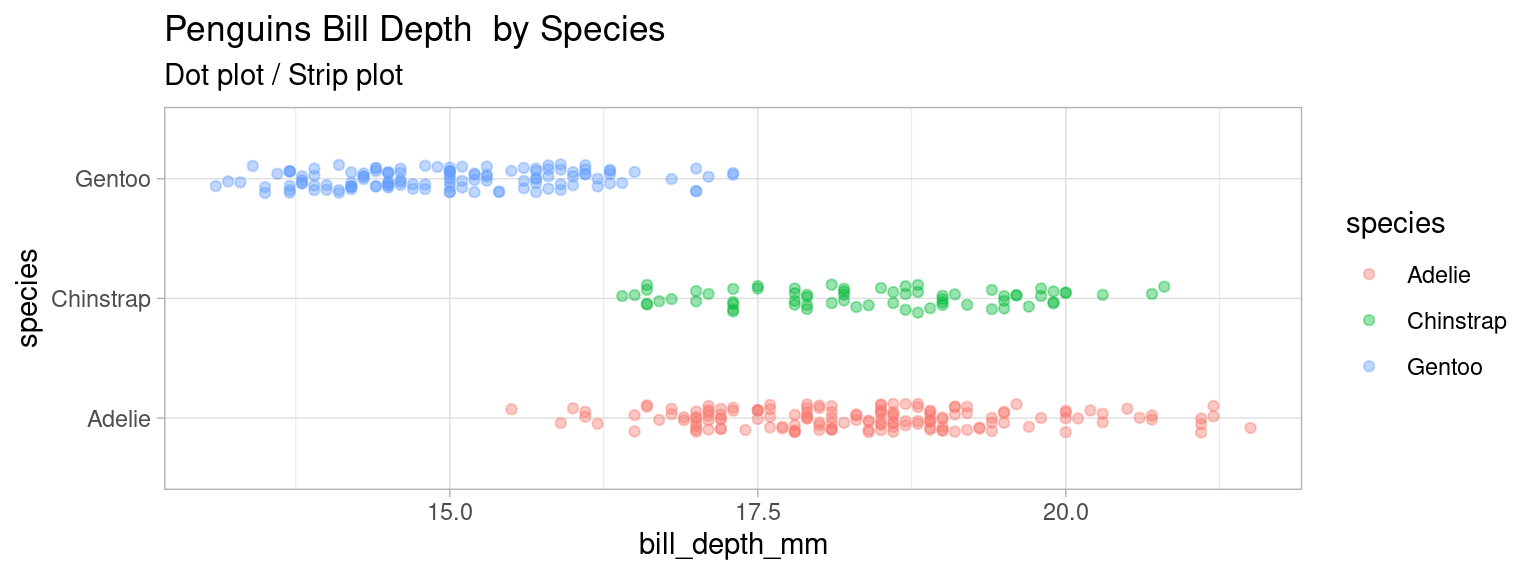
4.0.9 Dot Strip Plot
Cette visualisation est pratique lorsque les individus sont nombreux. En jouant sur la transparence et le décalage artificiel des points (fonction geom_jitter à la place de geom_point()), on peut faire apparaître les zones de la plages de valeurs de la variable où se retrouvent un nombre notable d’individus.
Cela reste à mon avis moins parlant qu’une bonne vieille courbe de densité traditionnelle.
library(palmerpenguins)
mydata <- penguins
plot_dot <- ggplot(mydata)+
geom_jitter(aes(x= bill_depth_mm, y=species, color=species),width=0, height = 0.12, alpha=0.4)+
labs(title = "Penguins Bill Depth by Species", subtitle = "Dot plot / Strip plot")+
theme_light()
plot_dot